
Un nuevo factor de transcripción esencial para la transcripción del virus de la gripe
Cada año, las mutaciones incorporadas en el genoma del virus de la gripe hacen necesaria la revacunación anual y el control por parte de las autoridades sanitarias para evitar pandemias. Con el objetivo de encontrar inhibidores de la actividad viral, diferentes grupos de investigación en todo el mundo trabajan para conocer mejor el mecanismo de la replicación viral y definir los factores celulares necesarios que puedan ayudar a controlar la infección.
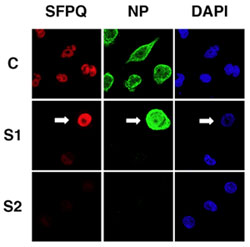 El virus de la gripe se
transcribe y replica en el núcleo de la célula infectada. Una vez que las
ribonucleoproteínas (RNPs) que forman el genoma viral llegan al núcleo, sirven
como molde para comenzar la transcripción primaria de los factores virales necesarios
para la replicación viral y la transcripción secundaria. Existe una estrecha
colaboración entre la maquinaria de transcripción viral y la celular. Para definir qué elementos celulares pueden ser importantes para el virus, en el CNB, El grupo
del Dr. Juan Ortín ha analizado la interacción de la RNA polimerasa viral con proteínas celulares. Su último trabajo, publicado en la revista PLoS
Pathogens, muestra el papel de una de estas proteínas, el factor de splicing SFPQ, en la infección
viral.
El virus de la gripe se
transcribe y replica en el núcleo de la célula infectada. Una vez que las
ribonucleoproteínas (RNPs) que forman el genoma viral llegan al núcleo, sirven
como molde para comenzar la transcripción primaria de los factores virales necesarios
para la replicación viral y la transcripción secundaria. Existe una estrecha
colaboración entre la maquinaria de transcripción viral y la celular. Para definir qué elementos celulares pueden ser importantes para el virus, en el CNB, El grupo
del Dr. Juan Ortín ha analizado la interacción de la RNA polimerasa viral con proteínas celulares. Su último trabajo, publicado en la revista PLoS
Pathogens, muestra el papel de una de estas proteínas, el factor de splicing SFPQ, en la infección
viral.
SFPQ es un factor que actúa tanto en la transcripción como en el splicing de mRNAs celulares. En relación a su papel en la infección causada por el virus de la gripe, los investigadores han observado que la disminución de los niveles de SFPQ afecta drásticamente a la producción de virus de la gripe, mientras que la multiplicación de otros virus como VSV o Adenovirus 5 son independientes de la presencia de SFPQ en la célula. Los autores han analizado en qué pasos del ciclo viral puede ser necesario SFPQ mediante el uso de infecciones en líneas celulares humanas o utilizando un sistema de replicones que permite analizar la replicación viral in vitro.
Estos experimentos han servido para demostrar de una forma muy elegante que la interacción de SFPQ con la RNA polimerasa viral es necesaria para producir mRNAs virales correctamente poliadenilados, un paso temprano del ciclo viral. La inhibición de esta interacción podría ser utilizada como diana para el desarrollo de antivirales específicos para el virus de la gripe.
Experimentos científicos en un ambiente de cero emisiones
En una jornada científica, investigadores de la Windpowered Antarctica's Challenge de la que formó parte Juan Pablo Albar, hablarán sobre los trabajos científicos que se llevan a cabo en este continente. A parte de demostrar la viabilidad de viajar en la meseta antártica con trineos movidos exclusivamente con la fuerza del viento, los científicos participantes han dedicado su tiempo a diversos experimentos que nos explicarán el próximo 27 de marzo de 2012.
El coronavirus de la gastroenteritis transmisible incorpora en su partícula viral proteínas necesarias para su replicación
¿Qué contiene una partícula viral? La respuesta más obvia sería el genoma del virus, imprescindible para su propagación. Las partículas virales contienen proteínas virales y celulares útiles en los primeros pasos de la replicación viral, además de otras que se han incorporado debido a una interacción con la propia célula infectada.
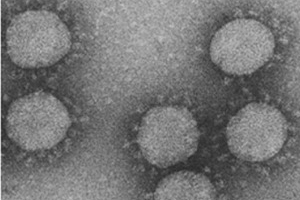 Hasta ahora, se aceptaba que los virus cuyo genoma está formado por RNA de cadena positiva no incorporaban proteínas implicadas en la replicación viral en sus viriones. Sin embargo, estudios más recientes están modificando esta hipótesis.
Hasta ahora, se aceptaba que los virus cuyo genoma está formado por RNA de cadena positiva no incorporaban proteínas implicadas en la replicación viral en sus viriones. Sin embargo, estudios más recientes están modificando esta hipótesis.
En esta dirección, el grupo supervisado por los doctores Luis Enjuanes y Fernando Almazán ha analizado mediante espectrometría de masas el contenido proteico de las partículas del coronavirus de la gastroenteritis transmisible (TGEV). Sus resultados, publicados en la revista Journal of Virology, muestran que en la partícula del TGEV no sólo están presentes proteínas estructurales del virus, sino también proteínas celulares como la calnexina, tubulina b5 o Hsp90AB ya identificadas en otros coronavirus como el causante del SARS (SARS-CoV) o de la bronquitis en aves.
El resultado más interesante es la identificación de la RNA polimerasa viral responsable de la replicación del virus (RdRp) dentro del virión. Mediante técnicas de microscopía electrónica de transmisión y microscopía confocal, se ha confirmado la presencia específica de la RdRp dentro de la partícula viral junto con otras proteínas virales implicadas en los procesos de replicación y transcripción viral como nsp2, nsp3 y nsp8.
Este nuevo hallazgo sugiere que la incorporación en los viriones de proteínas virales con un papel importante en los pasos iniciales de la replicación viral puede ser un mecanismo más general utilizado por diferentes familias de virus para producir una infección más eficiente.
Nogales A, Márquez-Jurado S, Galán C, Enjuanes L, Almazán F. Transmissible Gastroenteritis Coronavirus RNA-Dependent RNA Polymerase and Nonstructural Proteins 2, 3, and 8 Are Incorporated into Viral Particles. J Virol. 2012 Jan;86(2):1261-6.
Carmen Vela preside la entrega de los premios Biogen Idec
El próximo jueves, 15 de marzo, la secretaria de Estado de Investigación, Desarrollo e Innovación, Carmen Vela, entregará en la sede del Consejo Superior de Investigaciones Científicas (CSIC) los premios Fundación Biogen Idec para Jóvenes Investigadores Españoles. Estos galardones se iniciaron como una colaboración entre la Fundación Biogen Idec y el Centro Nacional de Biotecnología del CSIC y pretenden reconocer el mejor trabajo de investigación básica desarrollado por jóvenes científicos en las áreas de Neurología e Inmunología.
 La sexta edición premia en el área de Neurología la labor de Manuel López
Aranda, investigador de la Universidad de California, por su
contribución al descubrimiento del papel funcional de una población de
neuronas asociada a la formación de la memoria visual. En el área de
Inmunología, el reconocimiento por parte del jurado ha sido para la
investigadora Laura Belver, del Centro Nacional de Investigaciones
Cardiovasculares, por la novedad y las implicaciones terapéuticas que
tiene la demostración del papel esencial que juegan los micro-ARNs en la
diferenciación terminal de los linfocitos B, la generación de
auto-anticuerpos y el mantenimiento de la tolerancia inmunológica.
La sexta edición premia en el área de Neurología la labor de Manuel López
Aranda, investigador de la Universidad de California, por su
contribución al descubrimiento del papel funcional de una población de
neuronas asociada a la formación de la memoria visual. En el área de
Inmunología, el reconocimiento por parte del jurado ha sido para la
investigadora Laura Belver, del Centro Nacional de Investigaciones
Cardiovasculares, por la novedad y las implicaciones terapéuticas que
tiene la demostración del papel esencial que juegan los micro-ARNs en la
diferenciación terminal de los linfocitos B, la generación de
auto-anticuerpos y el mantenimiento de la tolerancia inmunológica.
En su dictamen el jurado de los premios Biogen Idec ha destacado la calidad de los trabajos recibidos, así como la de los curricula de los participantes, “lo cual es una muestra más del altísimo nivel de la investigación conseguido por los jóvenes investigadores españoles”.
En el acto de entrega participarán el vicepresidente de Investigación Científica y Técnica del CSIC, Antonio Figueras, y el investigador del departamento de Inmunología y Oncología del Centro Nacional de Biotecnología (CSIC), Carlos Martínez, que impartirá la lección magistral "Impacto de la Biología en la Medicina del Siglo XX".
Nuevo mecanismo de formación y transporte del ARN mensajero
Fruto de la colaboración con los grupos de Andrzej Dziembowski en la Academia Polaca de Ciencias y del Dr. Andrés Aguilera de la Universidad de Sevilla, el grupo de José María Valpuesta acaba de publicar en la revista EMBO Journal la estructura de un complejo de proteínas esenciales para la formación del ARN mensajero y su transporte desde el núcleo al citoplasma. Sus resultados revelan un nuevo mecanismo por el que la formación de este complejo proteico reconoce a los ácidos nucleicos y se une a ellos.
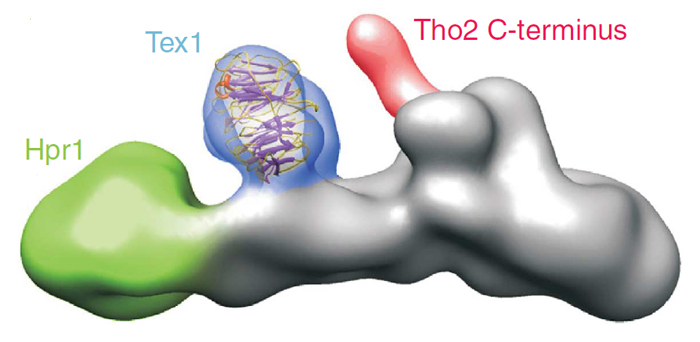
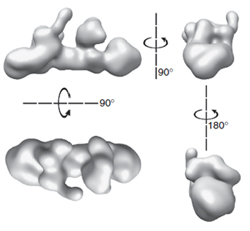 Como la transcripción del ADN tiene lugar en el núcleo de la célula y los ribosomas que forman las proteínas se encuentra en el citoplasma, un paso esencial para la expresión de cualquier gen es el transporte del ARN mensajero desde el núcleo hasta el citoplasma. Un proceso que se conoce desde hace bastante tiempo, pero cuyos detalles no se han investigado del todo.
Como la transcripción del ADN tiene lugar en el núcleo de la célula y los ribosomas que forman las proteínas se encuentra en el citoplasma, un paso esencial para la expresión de cualquier gen es el transporte del ARN mensajero desde el núcleo hasta el citoplasma. Un proceso que se conoce desde hace bastante tiempo, pero cuyos detalles no se han investigado del todo.
Se ha descrito que el complejo de proteínas THO está directamente relacionado con la transcripción, formación y transporte del ARN mensajero. Sin embargo, todavía no se ha descrito de forma clara ni su estructura ni su mecanismo de acción. El principal motivo es principalmete que este grupo de proteínas es tan poco soluble que ha sido imposible purificarlas en cantidades suficientes como para estudiarlo mediante cristalografía de rayos X.
En el Centro Nacional de Biotecnología del CSIC, el grupo de José María Valpuesta en colaboración con el grupo polaco, ha sido capaz de producir estas proteínas en suficiente cantidad y pureza como para poder ser analizadas mediante microscopía electrónica. De este modo, usando esta técnica en combinación diferentes sistemas de marcaje de proteínas, han sido capaces de elaborar un modelo a nivel atómico. En contra de lo que se creía hasta ahora, con su reconstrucción tridimensional han descubierto que el complejo no tiene cuatro sino cinco proteínas distintas. Y todo parece indicar que entre las funciones de este nuevo componente estaría la de unirse a otras proteínas que intervienen en el procesado, empaquetamiento y transporte del ARN mensajero.
Además, los tres grupos involucrados en el trabajo han sido capaces de identificar y localizar la zona de este complejo que se une de forma directa a los ácidos nucleicos, que como explica Valpuesta "es una región desestructurada que se encuentra en una de las proteínas del complejo". La relevancia de esta unión se pone de manifiesto por el hecho de que si se ve alterada se llega a producir un alto grado de inestabilidad genómica.
Reconstructing the essential bacterial cell cycle machinery
Miguel Vicente organiza en el Real Sitio de San Ildefonso el EMBO Workshop Reconstructing the essential bacterial cell cycle machinery. Del 16 al 19 de septiembre de 2012, los participantes tendrán la oportunidad de debatir los avances en la construcción de maquinarias bacterianas en ausencia de células. Los ponentes se centrarán especialmente en los componentes bacterianos implicados en procesos esenciales como la citocinesis, la segregación de los cromosomas y la síntesis de la pared bacteriana.
El CNIO y el CNB aúnan esfuerzos para almacenar y distribuir líneas de ratones a la comunidad científica
El Centro Nacional de Biotecnología del CSIC (CNB) acaba de firmar un acuerdo que permitirá almacenar y distribuir embriones y esperma de los ratones modificados genéticamente que se generan en el Centro Nacional de Investigaciones Oncológicas (CNIO). A través del European Mouse Mutant Archive (EMMA), los investigadores del CNIO podrán conservar en el CNB sus líneas de ratones. De este modo están disponibles para la investigación de cualquier científico interesado.
 Desde que en enero de 2009 el CNB se convirtiera en el nodo español de EMMA bajo la dirección de Lluís Montoliu, la comunidad científica española ha tenido más fácil el acceso libre a todas aquellas líneas de ratones transgénicos, knockouts o mutantes de interés para la investigación biomédica. A las líneas ya existentes se sumarán ahora aquellas provenientes del trabajo de los investigadores del CNIO, aumentando así la disponibilidad de estudiar la función de multitud de genes con interés en el campo de la oncología.
Desde que en enero de 2009 el CNB se convirtiera en el nodo español de EMMA bajo la dirección de Lluís Montoliu, la comunidad científica española ha tenido más fácil el acceso libre a todas aquellas líneas de ratones transgénicos, knockouts o mutantes de interés para la investigación biomédica. A las líneas ya existentes se sumarán ahora aquellas provenientes del trabajo de los investigadores del CNIO, aumentando así la disponibilidad de estudiar la función de multitud de genes con interés en el campo de la oncología.
Al ser un proyecto financiado por la Comisión Europea, la congelación y mantenimiento de ratones en condiciones EMMA es gratuita y el investigador sólo se debe responsabilizarse de los costes de traslado de sus modelos animales al nodo EMMA correspondiente (el CNB en Madrid, en este caso). Esto implica el acceso libre de los modelos animales depositados por la comunidad científica. No obstante, pueden asociarse a cada línea criopreservada en EMMA documentos que protejan la propiedad intelectual de los ratones depositados y puede solicitarse un periodo de hasta 2 años durante los cuales la línea está depositada en EMMA pero no aparece todavía en las bases de datos.
El nodo EMMA incluye la utilización de aisladores de jaulas de ratón, para permitir la recepción segura de ratones cuyo informe sanitario sea distinto al de los ratones alojados en el animalario del CNB. Se pretende con ello universalizar la criopreservación de líneas de ratón y ponerla a disposición de todos los investigadores interesados, con independencia de las características del animalario de origen.
Cryo X-ray nano-tomography of vaccinia virus infected cells
Chichón FJ, Rodríguez MJ, Pereiro E, Chiappi M, Perdiguero B, Guttmann P, Werner S, Rehbein S, Schneider G, Esteban M, Carrascosa JL.
 We have performed full-field cryo X-ray microscopy in the water window photon energy range on vaccinia virus (VACV) infected cells to produce tomographic reconstructions. PtK2 cells were infected with a GFP-expressing VACV strain and frozen by plunge fast freezing. The infected cells were selected by light fluorescence microscopy of the GFP marker and subsequently imaged in the X-ray microscope under cryogenic conditions.
We have performed full-field cryo X-ray microscopy in the water window photon energy range on vaccinia virus (VACV) infected cells to produce tomographic reconstructions. PtK2 cells were infected with a GFP-expressing VACV strain and frozen by plunge fast freezing. The infected cells were selected by light fluorescence microscopy of the GFP marker and subsequently imaged in the X-ray microscope under cryogenic conditions.Tomographic tilt series of X-ray images were used to yield three-dimensional reconstructions showing different cell organelles (nuclei, mitochondria, filaments), together with other structures derived from the virus infection. Among them, it was possible to detect viral factories and two types of viral particles related to different maturation steps of VACV (immature and mature particles), which were compared to images obtained by standard electron microscopy of the same type of cells. In addition, the effect of radiation damage during X-ray tomographic acquisition was analyzed. Thin sections studied by electron microscopy revealed that the morphological features of the cells do not present noticeable changes after irradiation.
Our findings show that cryo X-ray nano-tomography is a powerful tool for collecting three-dimensional structural information from frozen, unfixed, unstained whole cells with sufficient resolution to detect different virus particles exhibiting distinct maturation levels.
La microscopía de rayos X en la investigación biológica
El investigador del CNB José L. Carrascosa ha sido uno de los editores del número especial de la revista Journal of Structural Biology en el que se recogen las investigaciones más recientes sobre el uso en biología de la microscopía de rayos X. En este mismo número además, su grupo presenta sus últimos resultados aplicando esta técnica de microscopía al estudio de células infectadas por virus.
 Hoy en día, los investigadores se enfrentan al reto de resolver la
estructura de las células y sus componentes celulares en un estado lo más cercano posible al nativo. Aunque la utilidad de los rayos X para los biólogos se conoce desde hace ya bastante tiempo (miles de estructuras de proteínas y macromoléculas, incluida la del ADN, se han resuelto gracias a la cristalografía de rayos X), su uso para hacer microscopía no ha podido madurar hasta fechas muy recientes. Una serie de avances en óptica de rayos X, junto con la mejora de los métodos de preparación de células, han hecho posible construir microscopios de rayos X capaces de obtener imágenes de células completas a una resolución intermedia entre la microscopía electrónica y la microscopía óptica.
Hoy en día, los investigadores se enfrentan al reto de resolver la
estructura de las células y sus componentes celulares en un estado lo más cercano posible al nativo. Aunque la utilidad de los rayos X para los biólogos se conoce desde hace ya bastante tiempo (miles de estructuras de proteínas y macromoléculas, incluida la del ADN, se han resuelto gracias a la cristalografía de rayos X), su uso para hacer microscopía no ha podido madurar hasta fechas muy recientes. Una serie de avances en óptica de rayos X, junto con la mejora de los métodos de preparación de células, han hecho posible construir microscopios de rayos X capaces de obtener imágenes de células completas a una resolución intermedia entre la microscopía electrónica y la microscopía óptica.
Junto con Robert M. Glaeser de la University of California, Carrascosa ha seleccionado una serie de trabajos de grupos muy importantes a nivel internacional en el panorama de la biología estructural a nivel celular, en los que se desarrollan y aplican los avances más recientes en el campo de la microscopía de rayos X. Todos ellos tienen en común la combinación de esta técnica con otras que ayudan enormemente a mejorar los resultados obtenidos.
Por ejemplo, la combinación con los microscopios ópticos permite seleccionar las células más adecuadas para su estudio en profundidad con la microscopía de rayos X. Una selección previa que, a parte de suponer un ahorro de tiempo y esfuerzo, asegura al investigador que los resultados que obtiene son los correctos.
También se puede aprovechar la gran sensibilidad que ofrece la fluorescencia de rayos X para detectar y localizar cantidades realmente pequeñas de iones metálicos en el interior de las células. Algo que en el campo de la medicina es de enorme ayuda para poder estudiar las causas de diversas enfermedades metabólicas.
Gracias a la mejora de las ópticas de rayos X y al uso de métodos tomográficos de reconstrucción tridimensional, se ha podido desarrollar la tomografía de rayos X de células congeladas. Esta técnica permite obtener imágenes de células en su estado natural con una resolución de hasta 30 nm. Esto se traduce en imágenes en las que dentro de las células se pueden observar los virus que las infectan.
El grupo dirigido por Carrascosa (en el que han jugado un papel clave Javier Chichón y María José Rodríguez), utilizó unos virus especiales creados en colaboración con el grupo de Mariano Esteban, también del CNB, que permitía su detección por fluorescencia. Usando esta aproximación experimental, han sido capaces de detectar las zonas donde se ensamblan los virus, así como su ruta de maduración dentro de las células completas.
Con las células congeladas, utilizaron la microscopía de rayos X para ir obteniendo imágenes de ellas a distintos ángulos. Una vez procesada mediante métodos de reconstrucción tomográfica tridimensional toda la información obtenida, se han podido generar volúmenes tridimensionales de los virus que se encontraban dentro de las células. Unas imágenes de tal calidad y resolución que les ha permitido diferenciar dentro de la propia célula las distintas fases del proceso de maduración del virus.
La investigación sobre Enfermedades Raras en el Centro Nacional de Biotecnología del CSIC
Existen multitud de laboratorios que dedican sus esfuerzos a enfermedades como el cáncer, la diabetes, la gripe o el sida. Sin embargo, existen más de 7.000 patologías que afectan a menos de 1 de cada 2.000 personas y de las que probablemente nunca hayamos oído hablar. A ellas también dedican sus esfuerzos los científicos del CNB.
 En este Día Mundial de las Enfermedades Raras el científico del CNB Lluís Montoliu explica que "tomadas individualmente, cada enfermedad rara puede afectar a pocos o muy pocos pacientes". Por ejemplo, todos los tipos conocidos de albinismo afectan globalmente a una de cada 17.000 personas.
En este Día Mundial de las Enfermedades Raras el científico del CNB Lluís Montoliu explica que "tomadas individualmente, cada enfermedad rara puede afectar a pocos o muy pocos pacientes". Por ejemplo, todos los tipos conocidos de albinismo afectan globalmente a una de cada 17.000 personas.
Mucho menos frecuente una de las enfermedades sobre las que investiga Inés Antón. Tan sólo se conoce un paciente, aunque ya han sido capaces de saber que está causada por la deficiencia en una proteína llamada WIP. Y en entender cómo regula el desarrollo neuronal esta proteína, junto a otra causante del Sindrome de Wiskott Aldrich (una inmunodeficiencia también clasificada como enfermedad rara), trabaja Antón desde que se incorporara al CNB en 2007 .
Pero el hecho de que una enfermedad sea rara, no implica que no cause problemas serios por los que preocuparse. Como nos explica Montoliu, "globalmente las enfermedades raras son un problema sanitario relevante en cualquier país". De hecho, todos los años aparecen convocatorias de proyectos de investigación que pretenden ayudar a los científicos a mejorar tanto el diagnóstico como el tratamiento de unas enfermedades que, por muy raras que sean, afectan a casi tres millones de españoles.
Precisamente a otra de estas enfermedades, el albinismo, dedica Montoliu sus esfuerzos. Principalmente, a vencer las trabas que encuentran lo padres y los médicos para poder diagnosticarla correctamente y a tiempo. Para ello han desarrollado un kit para la detección de la mutaciones causantes de esta enfermedad.
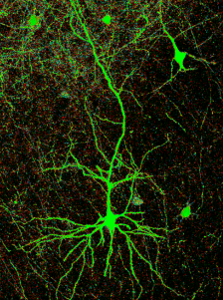 También en el campo de la neurociencias, el trabajo de José Ramón Naranjo pretende comprender mejor los mecanismos de la enfermedad de Huntington, un trastorno genético hereditario que provoca una pérdida de la coordinación motora y acaba provocando demencia. Descrita por primera vez en 1872, actualmente José Jesús Fernández aplica la experiencia de su laboratorio del CNB en tomografía electrónica para estudiar las alteraciones en la arquitectura subcelular de las neuronas afectadas por esta enfermedad.
También en el campo de la neurociencias, el trabajo de José Ramón Naranjo pretende comprender mejor los mecanismos de la enfermedad de Huntington, un trastorno genético hereditario que provoca una pérdida de la coordinación motora y acaba provocando demencia. Descrita por primera vez en 1872, actualmente José Jesús Fernández aplica la experiencia de su laboratorio del CNB en tomografía electrónica para estudiar las alteraciones en la arquitectura subcelular de las neuronas afectadas por esta enfermedad.
Afortunadamente, parece que estas patologías están siendo cada vez más investigadas gracias a la financiación de gobiernos y fundaciones. Algo totalmente necesario si queremos que en un futuro el acceso a los recursos sociosanitarios para casi tres millones de españoles sea mucho mas fácil que hoy en día.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.







