
Ignacio Cirac imparte la charla inaugural del XVII Ciclo de Seminarios del CNB
La conferencia inaugural del XVII Ciclo de Seminarios del Centro Nacional de Biotecnología del CSIC será impartida por el físico español Ignacio Cirac. Unos de los pioneros de la computación cuántica, fue galardonado en 2006 con el Premio Príncipe de Asturias de Investigación Científica y Técnica.
La conferencia del próximo viernes 28 de septiembre a las 12:00, Computers in the 21st century and beyond, será retransmitida por en directo vía streaming en la siguiente url:
http://www.cnb.csic.es/retransmisiones/Cirac2012.html
El XVII Ciclo de Seminarios consiste en una serie de conferencias de 13 magníficos científicos que tendrán lugar a lo largo del curso 20121 - 2013 los viernes a las 12:00 en el Salón de Actos del Centro Nacional de Biotecnología del CSIC.
III ProteoRed-ISCIII Protein Microarrays Course
Organizado por Manuel Fuentes, Concha Gil y el científico del CNB Juan Pablo Albar, los días 18 y 19 de octubre de 2012 se celebra en la Universidad Complutense de Madrid el III ProteoRed-ISCIII Protein Microarrays Course.
El curso se centra en un repaso a los conocimientos más actuales en el campo de Protein Microarrays, siendo una magnífica oportunidad para mejorar la competitividad de la unidades españolas de proteómica.
XX Workshop Avances en Biología Molecular por Jóvenes Investigadores en el Extranjero
Al cumplirse veinte años, además de que los jóvenes posdoctorales en el extranjero den a conocer su trabajo entre la comunidad científica española, científicos participantes en años anteriores y establecidos actualmente en España nos darán su visión de cómo ha cambiado la ciencia española en estos últimos 20 años.
Las jornadas finalizarán con una mesa redonda en la que se comentarán las distintas posibilidades de reincorporación al sistema científico español (empresas, universidades, hospitales y centros de investigación).
9:00 Carlos Martínez-Alonso: Welcome and presentation
Microbiology, Virology and Structural Biology session
(chair: Amelia Nieto)
09:15 Teresa del Peso
Pr – a model σ70-promoter for deciphering signal-integration mechanisms
Principal Investigator: Victoria Shingler, Department of Molecular Biology. Umeå University, Umeå (Sweden)
09:30 José Antonio Escudero
Surprises in the integron: DNA recombination is not that simple
Principal Investigator: Didier Mazel, Unité de Plasticité du Génome Bactérien. Institut Pasteur, Paris (France)
09:45 Diana Munera
Deciphering the virulence mechanisms of the E. coli O104:H4 German outbreak strain
Principal Investigator: Matt Waldor, Harvard Medical School, Boston (USA)
10:00 Ángel Rivera Calzada
Subnanometer resolution structure of a bacterial type IV secretion core complex
Principal Investigator: Gabriel Waksman, Birkbeck College-University College of London, London (UK)
10:15 Louis Chonco Jiménez
New vaccines strategies against HIV
Principal Investigator: Behazine Combadière, INSERM U945. Hôpital Pitié-Salpêtrière. Paris (France)
10:30 José Ignacio Garzón
PrePPI: a new database of protein-protein interactions with structural information
Principal Investigator: Barry Honig, Columbia University, New York (USA)
10:45 Patricia Resa Infante
The importin-α7 gene is a determinant of viral cell tropism in the murine lung
Principal Investigator: Gülsah Gabriel, Leibniz Institute for Experimental Virology, Hamburg (Germany)
(Coffee break)
Plant Biology session
(chair: Vicente Rubio)
11:30 Andrea Sánchez-Vallet
Ultra-high affinity chitin binding by intrachain LysM dimerization in the secreted fungal effector Ecp6
Principal Investigator: Bart Thomma, Laboratory of Phytopathology, Wageningen University (The Netherlands)
11:45 Amaury de Montaigu
Molecular regulation of diurnal rhythms by natural genetic variants
Principal Investigator: George Coupland, Max Planck Institute for Plant Breeding Research (Germany)
12:00 Maida Romera-Branchat
Characterization of a bZIP transcription factor involved in floral transition of Arabidopsis thaliana
Principal Investigator: George Coupland, Max Planck Institute for Plant Breeding Research (Germany)
12:15 Pilar Bustos-Sanmamed
Effects on transgene and endogenous siRNA biogenesis in Medicago truncatula rdr6 mutant
Principal Investigator: Martin Crespi, Institut des Sciences du Végétal, Gif-sur-Yvette (France)
20th Anniversary session
(chair: Mariano Esteban) 12:30
Dra. Margarita del Val, Centro de Biología Molecular 'Severo Ochoa', Madrid
Dr. Vicente Andrés, Centro Nacional de Investigaciones Cardiovasculares, Madrid
Dr. José Javier Lucas, Centro de Biología Molecular 'Severo Ochoa', Madrid
Dra. Carmen Simón, Centro Nacional de Biotecnología del CSIC, Madrid
Dr. Guillermo Montoya, Centro Nacional de Investigaciones Oncológicas
(Lunch)
Cellular and Molecular Biology, Immunology and Oncology session
(chair: Mario Mellado)
15:00 María Jiménez
Regulation of autophagy by the Hedgehog signalling pathway
Principal Investigator: David Rubinsztein, Cambridge Institute for Medical Research, Cambridge (UK)
15:15 Virginia Castilla Llorente
Mammalian GW220/TNGW1 is essential for the formation of GW/P bodies containing miRISC
Principal Investigator: Jidong Liu, Memorial Sloan-Kettering Cancer Center, New York (USA)
15:30 David Landeira
Jarid2 is required for gene priming and pluripotency of embryonic stem cells
Principal Investigator: Amanda Fisher, Medical Research Council, Clinical Science Centre, London (UK)
15:45 Nacho Molina
mRNA synthesis and degradation kinetics around the circadian cycle: transcriptional and post-transcriptional regulation
Principal Investigator: Felix Naef, Swiss Institute of Bioinformatics, EPFL, Lausanne (Switzerland)
16:00 Maria N. Navarro
From signal transduction networks to cell reprogramming: insights from phosphoproteomics in T cells
Principal Investigator: Doreen A. Cantrell, College of Life Sciences, University of Dundee, Scotland (UK)
16:15 Miguel Murillo
Role of Ras-PI3K interaction in tumor processes
Principal Investigator: Julian Downward, London Research Institute, Cancer Research UK, London (UK)
(Coffee break)
Incorporación al sistema científico español después de tu etapa en el extranjero
(chair: José María Valpuesta) 17:00
Dña. Carmen Vela Olmo, Secretaria de Estado de Investigación, Desarrollo e Innovación
Dr. Enrique Samper, Presidente de NIM Genetics
Dr. Guillermo Sellers, Director Científico de Investigación de Biogen España
Dra. María A. Blasco, Directora del Centro Nacional de Investigaciones Oncológicas
Dr. Francisco Sánchez-Madrid, Director Científico del Instituto de Investigación Sanitaria La Princesa
(Turrón y cava)
Organizadores:
Domingo F. Barber
Lourdes Planelles
Yolanda R. Carrasco
Colaboradores:
Fernando Almazán, Inés Antón, Silvia Ayora, Andrea Chini, Francisco J. Iborra, Alfonso Mora, Marta Nieto, Juan Poyatos y Carmen San Martín

Publican las conclusiones para prevenir otro brote de E. coli como el de 2011 en Alemania
Tras el brote de E. coli que afectó a Alemania y posteriormente a Francia la primavera del año pasado, la Academia Europea de Microbiología reunió el pasado diciembre en París a un grupo de expertos a nivel mundial para analizar lo sucedido y proponer estrategias para evitar o al menos minimizar los efectos de otros posibles brotes. En la revista científica EMBO Molecular Medicine y coordinado por el investigador del Centro Nacional de Biotecnología del CSIC Miguel Vicente, se publica ahora un artículo que recoge las conclusiones de la reunión.
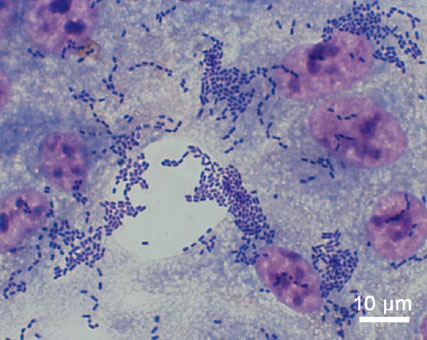 A pesar de la rapidez con la que se secuenció el genoma de la cepa de E. coli causante del brote, la mal llamada en España crisis de los pepinos se saldó con la muerte de 54 personas, en su mayoría ciudadanos alemanes, y una enormes pérdidas económicas en varios países, entre ellos España. Pese a todos los esfuerzos por identificarlo, no se tiene certeza de dónde ni cómo se originó el brote epidémico y solo existen explicaciones circunstanciales. Lamentablemente, un brote similar puede volver a darse en cualquier momento por lo que los científicos de la Academia Europea de Microbiología señalan una serie de recomendaciones para minimizar en lo posible sus efectos.
A pesar de la rapidez con la que se secuenció el genoma de la cepa de E. coli causante del brote, la mal llamada en España crisis de los pepinos se saldó con la muerte de 54 personas, en su mayoría ciudadanos alemanes, y una enormes pérdidas económicas en varios países, entre ellos España. Pese a todos los esfuerzos por identificarlo, no se tiene certeza de dónde ni cómo se originó el brote epidémico y solo existen explicaciones circunstanciales. Lamentablemente, un brote similar puede volver a darse en cualquier momento por lo que los científicos de la Academia Europea de Microbiología señalan una serie de recomendaciones para minimizar en lo posible sus efectos.
- La cepa O104:H4 muestra una sorprendente capacidad de adhesión al intestino y produce una toxina que provoca graves daños renales. Estos efectos por ahora sólo se pueden paliar eliminando la toxina mediante diálisis y manteniendo el equilibrio de electrolitos en sangre.
- Algunos antibióticos como meropenem, azitromicina o tigeciclina podrían utilizarse ya que la cepa es sensible a ellos. Otros antibióticos como la ciprofloxacina han de usarse con mucha precaución pues en dosis no inhibitorias pueden provocar un aumento en la producción de la toxina.
- Un anticuerpo comercializado como Eculizumab puede disminuir los efectos negativos de la respuesta inmunitaria que se genera en el organismo contra las toxinas de esta cepa de E. coli, pero tiene el riesgo asociado de disminuir la propia capacidad de defensa del cuerpo.
- Los expertos recomiendan que las autoridades sanitarias dispongan de procedimientos para que en este tipo de brotes el genotipado de las bacterias causantes se haga a la mayor brevedad posible. De el depende tanto el tratamiento como la eliminación de los focos de infección.
- Se hace asimismo hincapié en la necesidad de investigar a fondo los mecanismos por los que la toxina hace su efecto para así conseguir fármacos que impidan su acción en el riñón, y en la búsqueda de nuevos antibióticos que frenen la infección.
- Por último, proponen que se mejore la comunicación entre los científicos, los responsables políticos y los medios de difusión para que se afronten estos sucesos con una base científica fiable y adecuada a cada caso para minimizar los efectos sobre los ciudadanos y los sectores productivos.
Identificada una enzima clave en el inicio de la respuesta inmune
El grupo de Isabel Mérida acaba de identificar la enzima responsable del aumento del diacilglicerol en la sinapsis inmune, un paso esencial para que los linfocitos T puedan coordinar correctamente la respuesta inmunitaria.
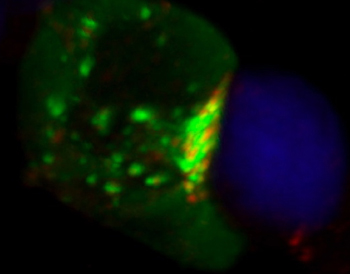 Desde su laboratorio del Centro Nacional de Biotecnología del CSIC, Mérida acaba de publicar un trabajo en el que la investigadora
postdoctoral Severine Gharbi identifica la enzima responsable del aumento de diacilglicerol en la zona de unión entre un linfocito T y un linfocito B. El aumento de este lípido en la llamada sinapsis inmune es necesario para que los linfocitos T puedan reconocer la proteína de los microorganismos contra los que dirigir su ataque.
Desde su laboratorio del Centro Nacional de Biotecnología del CSIC, Mérida acaba de publicar un trabajo en el que la investigadora
postdoctoral Severine Gharbi identifica la enzima responsable del aumento de diacilglicerol en la zona de unión entre un linfocito T y un linfocito B. El aumento de este lípido en la llamada sinapsis inmune es necesario para que los linfocitos T puedan reconocer la proteína de los microorganismos contra los que dirigir su ataque.
Además, marcando la llamada diacilglicerol kinasa ζ de tal modo que se pueda ver verde dentro del linfocito T, han observado cómo se concentra en la zona de unión al linfocito B (en azul). Una vez allí, produce el lípido necesario para que se reorganice la arquitectura de esta zona concreta de los linfocitos de cara a inicar la respuesta inmune.
- Gharbi SI, Rincón E, Ávila-Flores A, Torres-Ayuso P, Almena M, Cobos MA, Albar JP, Mérida I. Diacylglycerol kinase ζ controls diacylglycerol metabolism at the immunological synapse. Mol Biol Cell. 2011 Nov;22(22):4406-14.
p38γ y p38δ son claves en la respuesta inmunitaria
A pesar de las similitudes existentes entre las proteínas p38α, p38β, p38γ y p38δ, no están repartidas por igual en todos los tejidos y órganos y diversos compuestos químicos afectan a su actividad de manera distinta. Desde hace tiempo se sabe que las dos primeras participan en la respuesta inmune y en su laboratorio del Centro Nacional de Biotecnología del CSIC la Dra. Ana Cuenda estudia las funciones de las dos segundas, p38γ y p38δ.
 Esta misma semana su grupo ha publicado en la revista PNAS un trabajo en el que muestran que ambas proteínas desempeñan un papel fundamental en la respuesta inmune. En concreto en procesos inflamatorios como el choque séptico o la artritis reumatoide.
Esta misma semana su grupo ha publicado en la revista PNAS un trabajo en el que muestran que ambas proteínas desempeñan un papel fundamental en la respuesta inmune. En concreto en procesos inflamatorios como el choque séptico o la artritis reumatoide.
Para llegar a estas conclusiones tuvieron que generar unos ratones knockout que carecen de dichas proteínas. Con ellos, la investigadora postdoctoral Ana Risco ha comprobado que son menos sensibles al choque séptico, una respuesta inmune ante agentes infecciosos tan exagerada que acaba con la vida del ratón.
En su estudio han visto como en los macrófagos de sus ratones no se activan totalmente unas proteínas claves para la respuesta inmunitaria, ERK1 y ERK2. Por ello, se evita que se produzca choque séptico. Este resultado les hace pensar que otras enfermedades causadas por exceso de respuesta inmune tales como la artritis reumatoide pudieran también verse reducidas por la ausencia de las proteínas p38γ y p38δ.
Extrapolando estos datos a la personas, Cuenda explica que sus "resultados sugieren que terapias encaminadas a apagar la actividad de estas proteínas supondrían una mejora en los tratamientos actuales de enfermedades como la sepsis o la artritis reumatoide".
- Risco A, del Fresno C, Mambol A, Alsina-Beauchamp A, MacKenzie K, Yang H-T, Barber DF, Morcelle C, Arthur JSC, Ley SC, Ardavín C, Cuenda A. p38γ and p38δ kinases regulate TLR4-induced cytokine production by controlling ERK1/2 pathway activation. PNAS. 2012 Jun 25.
El citoesqueleto regula la expresión génica
La actina es una proteína forma el citoesqueleto de las células, participando en multitud de procesos como la elongación celular, el tráfico de proteínas a membrana o el movimiento de los orgánulos. Esta abundante proteína es prácticamente idéntica en todos los seres eucariotas, desde plantas a humanos, y suele tener funciones similares en todos los seres vivos donde se encuentra. El grupo de Antonio Leyva acaba de publicar en la revista Developmental Cell, que esta proteína es suficiente para alterar la expresión de los genes.
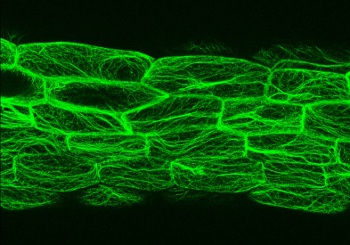 Estudiando la respuesta de las plantas a dos hormonas vegetales que regulan el crecimiento han visto que cambios en la estructura de los filamentos de actina tienen un efecto directo en la expresión de genes controlados por estas hormonas. Estos cambios son parte de procesos tan importantes como la respuesta de las plantas en la búsqueda de la luz, la capacidad de las raíces para responder a la gravedad y por tanto colonizar el suelo o la formación de raíces laterales.
Estudiando la respuesta de las plantas a dos hormonas vegetales que regulan el crecimiento han visto que cambios en la estructura de los filamentos de actina tienen un efecto directo en la expresión de genes controlados por estas hormonas. Estos cambios son parte de procesos tan importantes como la respuesta de las plantas en la búsqueda de la luz, la capacidad de las raíces para responder a la gravedad y por tanto colonizar el suelo o la formación de raíces laterales.
A pesar de que la actina se conoce desde hace más de 100 años, explica Leyva, "es una proteína prácticamente olvidada por la comunidad científica y nunca se le había atribuido un papel en la regulación transcripcional en plantas". De hecho, su trabajo es el primero que aporta evidencias genéticas de que los cambios en la configuración del citoesqueleto de actina tienen un efecto directo en la regulación de la expresión de genes mediados por hormonas regulando respuestas morfológicas vitales para las plantas.
La clave de este trabajo es un mutante del gen de la actina que mimetiza los mismos cambios en la expresión de genes que cuando se añaden las hormonas. Por ello, los investigadores concluyen que la actina no es un mero andamio en el interior celular sino que las hormonas parecen actuar mediante un efecto directo en la configuración del citoesqueleto.
- Lanza M, Garcia-Ponce B, Castrillo G, Catarecha P, Sauer M, Rodriguez-Serrano M, Páez-García A, Sánchez-Bermejo E, Tc M, Leo Del Puerto Y, Sandalio LM, Paz-Ares J, Leyva A. Role of actin cytoskeleton in brassinosteroid signaling and in its integration with the auxin response in plants. Dev Cell. 2012 Jun 12;22(6):1275-85.
Atacando a las bacterias donde más les duele
Investigadores del CSIC que participan en el proyecto de la Comisión Europea DIVINOCELL, han dado un paso más para saber cómo funciona una de las proteínas clave para que proliferen muchas bacterias, entre ellas la muy conocida Escherichia coli. Coordinado desde el Centro Nacional de Biotecnología por Miguel Vicente, el principal objetivo de este proyecto es el de encontrar nuevos antibióticos y, para ello, estudian las propiedades de FtsZ, la proteína que inicia el proceso por el que una bacteria se divide en dos.

Marisela Vélez, del Instituto de Catálisis y Petroleoquímica, también del CSIC, ha utilizado un microscopio de fuerza atómica, que permite observar con enorme detalle muestras sumergidas en una gota de líquido, para estudiar FtsZ. Sus observaciones han desvelado detalles muy importantes sobre cómo los polímeros de FtsZ, que son esenciales para la división de la bacteria, se curvan sobre una superficie de mica, llegando a formar círculos. Precisamente en la célula, FtsZ se localiza en el sitio donde se produce la división formando un anillo de mayores dimensiones que al estrecharse acaba por separar a las dos células.
Sobre la superficie de mica, los polímeros de FtsZ se estabilizan de forma transitoria, pero con el tiempo se van reduciendo de diámetro y los círculos se desvanecen. Esto ocurre porque las moléculas que componen el círculo se salen de él al azar, aunque las que se encuentran en los extremos lo hacen aún con mayor frecuencia. Según razonan los investigadores, si este proceso no existiese, los anillos difícilmente se estrecharían y la bacteria no se dividiría. El truco empleado para poder estudiar la formación y desaparición de los círculos ha sido utilizar unas condiciones bioquímicas que permiten ralentizar la separación de las moléculas de FtsZ que los componen. De este modo han podido estudiar este proceso con mayor precisión.
No cabe duda de que los compuestos que bloqueen por completo el proceso de formación o el de disgregación de los círculos de FtsZ inutilizarán el anillo de división en la bacteria impidiéndole su proliferación. Por ello, los investigadores quieren seguir colaborando con las empresas biotecnológicas que participan en el proyecto hasta encontrar un nuevo antibiótico.
En el trabajo, publicado en la revista de la Academia de Ciencias de los Estados Unidos ha requerido una notable integración de diversos conocimientos y técnicas. De hecho, en estas investigaciones han participado el grupo del Centro de Investigaciones Biológicas dirigido por Germán Rivas, científicos del Instituto de Ciencia de Materiales Nicolás Cabrera y el investigador del Departamento de Física Teórica de la Materia Condensada de la Universidad Autónoma de Madrid Pedro Tarazona.
- Mateos-Gil P, Paez A, Hörger I, Rivas G, Vicente M, Tarazona P, Vélez M. Depolymerization dynamics of individual filaments of bacterial cytoskeletal protein FtsZ. Proc Natl Acad Sci USA. 2012 May 22;109(21):8133-8.
MadrI+D premia el proyecto empresarial de Antonio Ramos
El proyecto empresarial del científico del CNB Antonio Ramos Service-Oriented Proteomics Technologies ha sido galardonado con uno de los premios MadrI+D. La ceremonia de entrega tendrá lugar hoy viernes 15 de junio a las 12:00h en el anfiteatro de la Casa de América de Madrid.
 Los premios madrI+D
se conceden a investigadores, inventores, emprendedores y autores de
blogs del sistema madrI+D, por la realización de una invención, proyecto
empresarial de base científico-tecnológica, proyecto europeo de
investigación liderado o difusión del conocimiento, atendiendo al
carácter innovador, mérito inventivo, potencial de desarrollo y
generación de ámbitos de comunicación en ciencia y tecnología.
Los premios madrI+D
se conceden a investigadores, inventores, emprendedores y autores de
blogs del sistema madrI+D, por la realización de una invención, proyecto
empresarial de base científico-tecnológica, proyecto europeo de
investigación liderado o difusión del conocimiento, atendiendo al
carácter innovador, mérito inventivo, potencial de desarrollo y
generación de ámbitos de comunicación en ciencia y tecnología.
Además de Ramos, otros cuatro científicos del CSIC han sido galardonados en esta octava edición.
Las patentes premiadas son: ‘Secuencia de nucleótidos péptidos GSE 24.2 de la disquerina inductores de la actividad telomerasa, procedimiento de obtención, composiciones terapéuticas’, cuya inventora principal es la profesora Rosario Perona de Instituto de Investigaciones Biomédicas ‘Alberto Solís’ (CSIC-UAM); y ‘Método para la identifiación de compuestos que inducen o inhiben estrés de retículo endoplásmico o estrés oxidativo’, de la doctora María Jesús Bullido del Centro de Biología Molecular Severo Ochoa (CSIC-UAM).
Por último, los premios de comunicación científica: blogs mi+d han sido otorgados a Antonio Figueras, vicepresidente del Consejo y profesor del Instituto de Investigaciones de Ciencias Marinas (CSIC), por ‘Ciencia Marina y otros asuntos’; y José Antonio López, director de cultura científica del Centro de Biología Molecular Severo Ochoa (CSIC-UAM).
Juan Luis Ramos, premio Rey Jaime I de Protección del Medio Ambiente
El jurado de los premios Rey Jaime I, formado por 22 premios Nobel, ha otorgado este año el galardón de Protección del Medio Ambiente al miembro del Comité Científico Asesor del CNB Juan Luis Ramos, por su trabajo sobre el uso de los microbios para aplicaciones medioambientales y biotecnológicas en la eliminación de contaminantes.
 Desde la Estación Experimental del Zaidín, Ramos ha contribuido en la lucha contra la contaminación medioambiental mediante el desarrollo de herramientas biológicas. Los compuestos a combatir son hidrocarburos aromáticos (benceno, tolueno, etilbenceno y xilenos), nitroaromáticos como el TNT, haloaromáticos como los PCBs y pesticidas como el lindano y el DDT. Su enfoque experimental incluye técnicas moleculares y el análisis bioquímico y genético de las rutas catabólicas, poniendo especial énfasis en las interacciones ADN/regulador, en experimentos en reactores y en la llamada rizorremediación de suelos.
Desde la Estación Experimental del Zaidín, Ramos ha contribuido en la lucha contra la contaminación medioambiental mediante el desarrollo de herramientas biológicas. Los compuestos a combatir son hidrocarburos aromáticos (benceno, tolueno, etilbenceno y xilenos), nitroaromáticos como el TNT, haloaromáticos como los PCBs y pesticidas como el lindano y el DDT. Su enfoque experimental incluye técnicas moleculares y el análisis bioquímico y genético de las rutas catabólicas, poniendo especial énfasis en las interacciones ADN/regulador, en experimentos en reactores y en la llamada rizorremediación de suelos.
Su grupo de investigación estudia también las interacciones entre plantas y microorganismos en la rizosfera con el objetivo de explotar los sistemas de expresión génica en bacterias para el desarrollo de sistemas de biocontrol, biodegradación y contención biológica de microorganismos recombinantes.
Desde el CNB queremos transmitirle nuestra más sincera enhorabuena.POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.






