 Adenovirus (AdV) capsid organization is considerably complex, not only
because of its large size (~950 Å) and triangulation number (pseudo T =
25), but also because it contains four types of minor proteins in
specialized locations modulating the quasi-equivalent icosahedral
interactions. Up until 2009, only its major components (hexon, penton,
and fiber) had separately been described in atomic detail. Their
relationships within the virion, and the location of minor coat
proteins, were inferred from combining the known crystal structures with
increasingly more detailed cryo-electron microscopy (cryoEM) maps.
Adenovirus (AdV) capsid organization is considerably complex, not only
because of its large size (~950 Å) and triangulation number (pseudo T =
25), but also because it contains four types of minor proteins in
specialized locations modulating the quasi-equivalent icosahedral
interactions. Up until 2009, only its major components (hexon, penton,
and fiber) had separately been described in atomic detail. Their
relationships within the virion, and the location of minor coat
proteins, were inferred from combining the known crystal structures with
increasingly more detailed cryo-electron microscopy (cryoEM) maps.
There was no structural information on assembly intermediates. Later on
that year, two reports described the structural differences between the
mature and immature adenoviral particle, starting to shed light on the
different stages of viral assembly, and giving further insights into the
roles of core and minor coat proteins during morphogenesis.
Finally, in 2010, two papers describing the atomic resolution structure
of the complete virion appeared. These reports represent a
veritable tour de force for two structural biology techniques: X-ray
crystallography and cryoEM, as this is the largest macromolecular
complex solved at high resolution by either of them. In particular, the
cryoEM analysis provided an unprecedented clear picture of the complex
protein networks shaping the icosahedral shell. Here I review these
latest developments in the field of AdV structural studies.
El grupo de Isabel Mérida acaba de identificar la enzima responsable del aumento del diacilglicerol en la sinapsis inmune, un paso esencial para que los linfocitos T puedan coordinar correctamente la respuesta inmunitaria.
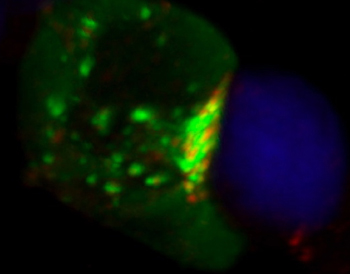 Desde su laboratorio del Centro Nacional de Biotecnología del CSIC, Mérida acaba de publicar un trabajo en el que la investigadora
postdoctoral Severine Gharbi identifica la enzima responsable del aumento de diacilglicerol en la zona de unión entre un linfocito T y un linfocito B. El aumento de este lípido en la llamada sinapsis inmune es necesario para que los linfocitos T puedan reconocer la proteína de los microorganismos contra los que dirigir su ataque.
Desde su laboratorio del Centro Nacional de Biotecnología del CSIC, Mérida acaba de publicar un trabajo en el que la investigadora
postdoctoral Severine Gharbi identifica la enzima responsable del aumento de diacilglicerol en la zona de unión entre un linfocito T y un linfocito B. El aumento de este lípido en la llamada sinapsis inmune es necesario para que los linfocitos T puedan reconocer la proteína de los microorganismos contra los que dirigir su ataque.
Además, marcando la llamada diacilglicerol kinasa ζ de tal modo que se pueda ver verde dentro del linfocito T, han observado cómo se concentra en la zona de unión al linfocito B (en azul). Una vez allí, produce el lípido necesario para que se reorganice la arquitectura de esta zona concreta de los linfocitos de cara a inicar la respuesta inmune.
- Gharbi SI, Rincón E, Ávila-Flores A, Torres-Ayuso P, Almena M, Cobos MA, Albar JP, Mérida I. Diacylglycerol kinase ζ controls diacylglycerol metabolism at the immunological synapse. Mol Biol Cell. 2011 Nov;22(22):4406-14.
Para poder seguir desarrollando la idea de sustituir a los antibióticos por virus que maten a las bacterias, en el Centro Nacional de Biotecnología del CSIC (CNB) estudian las proteínas que permiten a esos virus bacteriófagos anclarse sobre la superficie de las bacterias. Y acaban de publicar en la revista PNAS un avance realmente interesante, desvelando la estructura de una de estas proteínas.
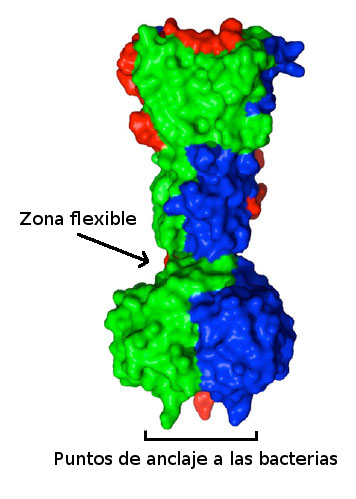 Cada bacteriófago se adhiere de forma muy específica a una especie concreta de bacteria, por lo que, en principio estos virus nunca podrían usarse de forma generalizada. Por ello, y como parte de un proyecto financiado por la Fundación Bill & Melinda Gates, en el grupo de Mark van Raaij trabajan sobre la idea de crear mediante mutaciones puntuales una gran variedad de bacteriófagos que puedan ser utilizados contra el tipo de bacteria que se desee. Su primer paso ha sido estudiar el mecanismo exacto por el que los bacteriófagos se colocan sobre la bacteria y se anclan a su membrana justo antes de empezar la destrucción de la misma.
Cada bacteriófago se adhiere de forma muy específica a una especie concreta de bacteria, por lo que, en principio estos virus nunca podrían usarse de forma generalizada. Por ello, y como parte de un proyecto financiado por la Fundación Bill & Melinda Gates, en el grupo de Mark van Raaij trabajan sobre la idea de crear mediante mutaciones puntuales una gran variedad de bacteriófagos que puedan ser utilizados contra el tipo de bacteria que se desee. Su primer paso ha sido estudiar el mecanismo exacto por el que los bacteriófagos se colocan sobre la bacteria y se anclan a su membrana justo antes de empezar la destrucción de la misma.
Analizando con precisión la estructura de las fibras mediante las que el bacteriófago T7 se une a las bacterias, Carmela García Doval y Mark van Raaij han comprobado que están formadas por tres unidades de una misma proteína. Mediante cristalografía de rayos X han determinado la existencia de un área justo antes de la zona que se une a las bacterias que dota a estas fibras de flexibilidad. Una flexibilidad que parece ser importante a la hora de que el virus se ancle correctamente sobre la pared bacteriana.
Gracias a la alta resolución de los datos obtenidos, han podido localizar con precisión las zonas concretas a través de las cuales se produce la unión entre el fago y la bacteria. Estos datos corroboran lo que venían indicando estudios previos de los aminoácidos que forman estas proteínas. Ahora, en su laboratorio del CNB, el grupo de van Raaij pretende generar bacteriófagos que contengan mutaciones aleatorias en las zonas que determinan su unión a las bacterias.
Con los miles de mutantes que planean obtener, tendrán que ir analizando la especificidad con la que se unen a las diferentes bacterias. Una vez que hayan detectado los mutantes que eliminan específicamente a las bacterias patógenas que les interesen, se producirán en grandes cantidades para ensayar su uso como posible tratamiento.
García-Doval C, van Raaij MJ. Structure of the receptor-binding carboxy-terminal domain of bacteriophage T7 tail fibers. Proc Natl Acad Sci USA. 2012 May 28.
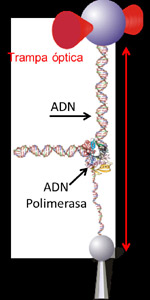
Manipulando moléculas individuales de la ADN polimerasa del virus Phi29, el equipo de Borja Ibarra ha conseguido cuantificar por primera vez el mecanismo de apertura del ADN utilizado por esta proteína. Publicado en la revista PNAS, este trabajo ayudará a desarrollar nanomotores sintéticos.
 Muchas de las proteínas del interior celular funcionan como auténticos motores moleculares capaces de trabajar eficientemente a escalas nanométricas (mil veces más pequeñas que la de la célula). Para ello, transforman la energía disponible en el interior celular en diminutas fuerzas (picoNewtons: la diez millonésima parte del peso de un miligramo) y desplazamientos (nanómetros: la millonésima parte de un milímetro). Unos de los motores moleculares mas sorprendentes son las ADN polimerasas, las proteínas encargadas de duplicar la doble hélice del ADN que son capaces de leer la composición de bases de cada una de las hebras del ADN e incorporar la base complementaria en cada posición. Sorprendentemente, la polimerasa del virus Phi29 puede avanzar a lo largo del ADN a una velocidad de 6.000 bases por minuto a la vez que abre la doble hélice de la molécula y avanza por ella. Un corredor de obstáculos con estas propiedades sería capaz de correr aproximadamente a 360 km/h e ir saltando vallas separadas por solo 1 metro ¡sin tirar ninguna! Estas sorprendentes propiedades y habilidades de estos nanomotores biológicos, han fascinado durante mucho tiempo a los biólogos moleculares y en los últimos años a una amplia comunidad de físicos e investigadores del área de la nanotecnología.
Muchas de las proteínas del interior celular funcionan como auténticos motores moleculares capaces de trabajar eficientemente a escalas nanométricas (mil veces más pequeñas que la de la célula). Para ello, transforman la energía disponible en el interior celular en diminutas fuerzas (picoNewtons: la diez millonésima parte del peso de un miligramo) y desplazamientos (nanómetros: la millonésima parte de un milímetro). Unos de los motores moleculares mas sorprendentes son las ADN polimerasas, las proteínas encargadas de duplicar la doble hélice del ADN que son capaces de leer la composición de bases de cada una de las hebras del ADN e incorporar la base complementaria en cada posición. Sorprendentemente, la polimerasa del virus Phi29 puede avanzar a lo largo del ADN a una velocidad de 6.000 bases por minuto a la vez que abre la doble hélice de la molécula y avanza por ella. Un corredor de obstáculos con estas propiedades sería capaz de correr aproximadamente a 360 km/h e ir saltando vallas separadas por solo 1 metro ¡sin tirar ninguna! Estas sorprendentes propiedades y habilidades de estos nanomotores biológicos, han fascinado durante mucho tiempo a los biólogos moleculares y en los últimos años a una amplia comunidad de físicos e investigadores del área de la nanotecnología.
Clásicamente, el funcionamiento de estos motores se ha estudiado en tubos de ensayo donde millones de polimerasas trabajan al mismo tiempo en una reacción no sincronizada. De esta manera, muchos de los detalles del funcionamiento intrínseco de cada polimerasa se pierden en el promediado final. En colaboración con los grupo de José María Valpuesta y José L. Carrascosa en el Centro Nacional de Biotecnología del CSIC, de Francisco J. Cao en la Universidad Complutense de Madrid y de Margarita Salas del Centro de Biología Molecular “Severo Ochoa”, Borja Ibarra ha utilizado en su laboratorio del Instituto Madrileño de Estudios Avanzados en Nanocienca (IMDEA Nanociencia) la técnica de las pinzas ópticas para atrapar y manipular moléculas individuales de la polimerasa del virus Phi29. De esta forma estos investigadores han podido seguir la actividad de una sola molécula de polimerasa mientras trabaja y se desplaza abriendo la doble hélice del ADN.
En su estudio han encontrando que este diminuto motor molecular es capaz de acoplar la energía térmica con la energía química derivada de la incorporación de nucleótidos en la ruptura de los puentes de hidrógeno que mantienen unidas las dos cadenas del ADN. De este modo, se llegan a ejercer sobre la doble cadena de ADN fuerzas superiores a los 10 pN (1010 veces su peso). Escalando la polimerasa al tamaño microscópico, un motor del tamaño del de un coche con la misma relación fuerza-masa sería capaz de ejercer fuerzas efectivas iguales al peso de 300 millones de toneladas métricas, o el peso de unos 700 portaaviones.
Hace tan solo unos años la posibilidad de estudiar los sistemas biológicos a nivel de moléculas individuales parecía una historia sacada de algún libro de ciencia ficción, pero la implementación de avanzadas técnicas de manipulación de moléculas individuales tanto en el IMDEA Nanociencia como en el Centro Nacional de Biotecnología del CSIC está permitiendo que estas historias se hagan realidad. El estudio de los motores moleculares biológicos con estas técnicas esta permitiendo por primera vez en España el establecimiento de una amplia red de colaboración interdisciplinar entre biofísicos, biólogos moleculares y físicos. Además estos nuevos descubrimientos permiten conocer mejor el funcionamiento interno de las células y harán posible diseñar en el futuro nanomáquinas sintéticas que emulen la ingeniosa y eficiente maquinaria molecular diseñada por la naturaleza.
Morin JA, Cao FJ, Lázaro JM, Arias-Gonzalez JR, Valpuesta JM, Carrascosa JL, Salas M, Ibarra B. Active DNA unwinding dynamics during processive DNA replication. Proc Natl Acad Sci USA. 2012 May 9.
En colaboración con IMDEA Nanociencia, el investigador del CNB Dr. Fernando Moreno ha organizado el 2º Workshop en Nanobiociencia. Tendrá lugar el próximo viernes 18 de Mayo en la sede del IMDEA Nanociencia, en el campus de la Universidad Autónoma de Madrid.
 Esta jornada científica, de un día de duración y con entrada libre, presenta una pequeña muestra de la biociencia de molécula individual que se realiza en España usando técnicas biofísicas como Microscopía de Fuerzas Atómicas (AFM), Pinzas Ópticas y Magnéticas, o Microscopía de Fluorescencia de Alta resolución.
Esta jornada científica, de un día de duración y con entrada libre, presenta una pequeña muestra de la biociencia de molécula individual que se realiza en España usando técnicas biofísicas como Microscopía de Fuerzas Atómicas (AFM), Pinzas Ópticas y Magnéticas, o Microscopía de Fluorescencia de Alta resolución.
El programa incluye cuatro sesiones con dos conferencias plenarias de investigadores extranjeros expertos en AFM y Pinzas Ópticas y contribuciones cortas de jóvenes biofísicos españoles.
Rodríguez-Beltrán J, Rodríguez-Rojas A, Guelfo JR, Couce A, Blázquez J.
DNA is constantly damaged by physical and chemical factors, including
reactive oxygen species (ROS), such as superoxide radical (O2−), hydrogen peroxide (H2O2)
and hydroxyl radical (•OH). Specific mechanisms to protect and repair
DNA lesions produced by ROS have been developed in living beings.
In Escherichia coli the SOS system, an inducible response activated to rescue cells from severe DNA damage, is a network that regulates the expression of more than 40 genes in response to this damage, many of them playing important roles in DNA damage tolerance mechanisms. Although the function of most of these genes has been elucidated, the activity of some others, such as dinF, remains unknown. The DinF deduced polypeptide sequence shows a high homology with membrane proteins of the multidrug and toxic compound extrusion (MATE) family. We describe here that expression of dinF protects against bile salts, probably by decreasing the effects of ROS, which is consistent with the observed decrease in H2O2-killing and protein carbonylation.
These results, together with its ability to decrease the level of intracellular ROS, suggests that DinF can detoxify, either direct or indirectly, oxidizing molecules that can damage DNA and proteins from both the bacterial metabolism and the environment. Although the exact mechanism of DinF activity remains to be identified, we describe for the first time a role for dinF.
El Dr. Lluís Montoliu, investigador científico del Centro Nacional de Biotecnología del CSIC, y coordinador del nodo español del Proyecto Europeo EMMA (European Mouse Mutant Archive), preside la organización del Cryopreservation Workshop en 2012 que se celebrará en Madrid, en la sede central del CSIC, los días 7 y 8 de mayo de 2012.
 Desde EMMA, esta reunión sobre criopreservación se plantea como un foro para discutir al más alto nivel y en profundidad, los últimos avances tecnológicos en la criopreservación de líneas de ratones. Estas técnicas incluyen la criopreservación de esperma y de embriones, métodos actualizados de fecundación in vitro (FIV) y técnicas relacionadas, como criopreservación de ovarios, inyección intracitoplasmática de esperma (ICSI) asistida por laser y piezoinyectores, transporte de material biológico congelado y cualquier otra técnica o reto logístico relevante para el funcionamiento normal de los bancos actuales de embriones y esperma.
Desde EMMA, esta reunión sobre criopreservación se plantea como un foro para discutir al más alto nivel y en profundidad, los últimos avances tecnológicos en la criopreservación de líneas de ratones. Estas técnicas incluyen la criopreservación de esperma y de embriones, métodos actualizados de fecundación in vitro (FIV) y técnicas relacionadas, como criopreservación de ovarios, inyección intracitoplasmática de esperma (ICSI) asistida por laser y piezoinyectores, transporte de material biológico congelado y cualquier otra técnica o reto logístico relevante para el funcionamiento normal de los bancos actuales de embriones y esperma.
Esta será una reunión limitada, con la asistencia prevista de 60 participantes, por invitación, que incluyen los mejores expertos del campo a nivel mundial. Esta reunión está copatrocinada por la International Society for Transgenic Technologies (ISTT) y 10 de sus miembros han sido seleccionados para asistir a partir de las solicitudes recibidas. Los participantes invitados son:
- de los EEUU: Rob Taft, Michael Wiles, Kent Lloyd, Jorge Sztein, Carlisle Landel, Peter Mazur;
- de Canadá: Marina Gertsenstein, Lauryl Nutter;
- de Latino-América: Martina Crispo;
- de Australia: Stuart Read, Sue Bath;
- de China: Xiang Gao;
- de Japón: Naomi Nakagata, Toru Takeo, Keiji Mochida, Atsuo Ogura;
- de Europa: Martin Fray, Pedro Moreira, Alan Hart, Xavier Warot, Jean Jaubert, Marcello Raspa, Sagrario Ortega y Belén Pintado.
La revista Structure acaba de publicar el trabajo del grupo de Cristina Risco en el que desarrollan un marcador clonable para la detección de forma individual de una o varias proteínas por microscopía electrónica.
 La microscopía electrónica ha contribuido más que ninguna otra metodología a nuestra comprensión de la arquitectura y organización celular. Sin embargo, la detección de proteínas en células a nivel ultraestructural sigue realizándose hoy en día mediante técnicas de inmunomarcaje que no tienen ni la sensibilidad ni la resolución que cabría esperar del uso de los microscopios electrónicos. De ahí que se siga intentando desarrollar nuevas técnicas que permitan identificar de forma directa y de manera cuantitativa las moléculas individuales de las proteínas en el denso entorno intracelular.
La microscopía electrónica ha contribuido más que ninguna otra metodología a nuestra comprensión de la arquitectura y organización celular. Sin embargo, la detección de proteínas en células a nivel ultraestructural sigue realizándose hoy en día mediante técnicas de inmunomarcaje que no tienen ni la sensibilidad ni la resolución que cabría esperar del uso de los microscopios electrónicos. De ahí que se siga intentando desarrollar nuevas técnicas que permitan identificar de forma directa y de manera cuantitativa las moléculas individuales de las proteínas en el denso entorno intracelular.
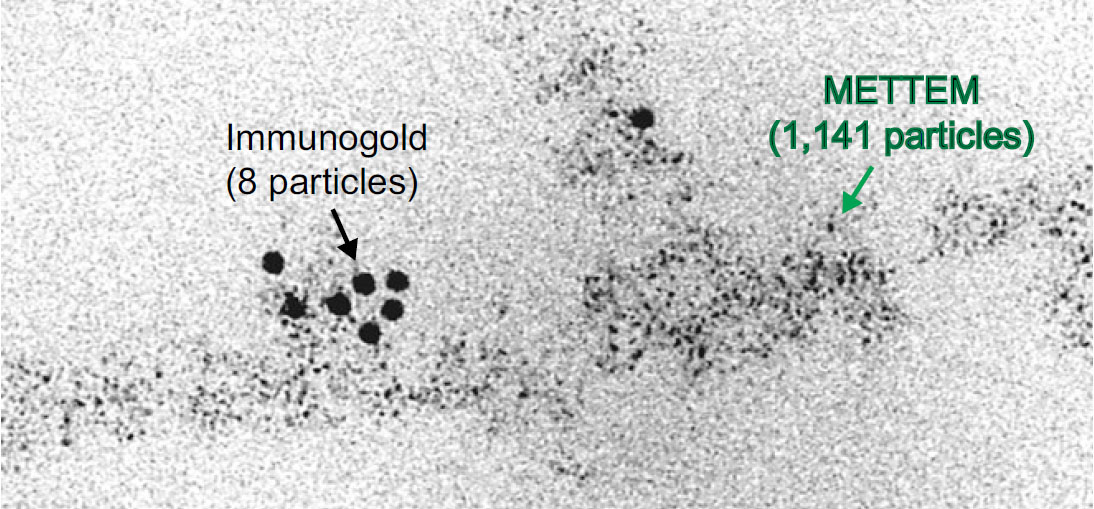
El nuevo marcador que ha patentado el CSIC junto con el método utilizado para su visualización denominado Metal-Tagging Transmision Electron Microscopy (METTEM), contiene la secuencia de una pequeña proteína que une metales y le permite formar nanopartículas de 1 nm fácilmente detectables con el microscopio electrónico. La propia Dra. Risco explica que este nuevo método "permite la detección de proteínas en células con gran especificidad, sensibilidad excepcional y resolución molecular".
Como se puede apreciar en la imagen, este marcador (METTEM) tiene una sensibilidad superior en varios órdenes de magnitud a la proporcionada por los anticuerpos en ensayos de inmunomarcaje (Immunogold). Por ello, presenta grandes perspectivas para la visualización y el estudio de las biomoléculas en su entorno celular nativo. La trascendencia en el campo de la microscopía electrónica es potencialmente similar a la alcanzada por las proteínas fluorescentes en microscopía óptica.
Miguel Vicente organiza en el Real Sitio de San Ildefonso el EMBO Workshop Reconstructing the essential bacterial cell cycle machinery. Del 16 al 19 de septiembre de 2012, los participantes tendrán la oportunidad de debatir los avances en la construcción de maquinarias bacterianas en ausencia de células. Los ponentes se centrarán especialmente en los componentes bacterianos implicados en procesos esenciales como la citocinesis, la segregación de los cromosomas y la síntesis de la pared bacteriana.
El Centro Nacional de Biotecnología del CSIC (CNB) acaba de firmar un acuerdo que permitirá almacenar y distribuir embriones y esperma de los ratones modificados genéticamente que se generan en el Centro Nacional de Investigaciones Oncológicas (CNIO). A través del European Mouse Mutant Archive (EMMA), los investigadores del CNIO podrán conservar en el CNB sus líneas de ratones. De este modo están disponibles para la investigación de cualquier científico interesado.
 Desde que en enero de 2009 el CNB se convirtiera en el nodo español de EMMA bajo la dirección de Lluís Montoliu, la comunidad científica española ha tenido más fácil el acceso libre a todas aquellas líneas de ratones transgénicos, knockouts o mutantes de interés para la investigación biomédica. A las líneas ya existentes se sumarán ahora aquellas provenientes del trabajo de los investigadores del CNIO, aumentando así la disponibilidad de estudiar la función de multitud de genes con interés en el campo de la oncología.
Desde que en enero de 2009 el CNB se convirtiera en el nodo español de EMMA bajo la dirección de Lluís Montoliu, la comunidad científica española ha tenido más fácil el acceso libre a todas aquellas líneas de ratones transgénicos, knockouts o mutantes de interés para la investigación biomédica. A las líneas ya existentes se sumarán ahora aquellas provenientes del trabajo de los investigadores del CNIO, aumentando así la disponibilidad de estudiar la función de multitud de genes con interés en el campo de la oncología.
Al ser un proyecto financiado por la Comisión Europea, la congelación y mantenimiento de ratones en condiciones EMMA es gratuita y el investigador sólo se debe responsabilizarse de los costes de traslado de sus modelos animales al nodo EMMA correspondiente (el CNB en Madrid, en este caso). Esto implica el acceso libre de los modelos animales depositados por la comunidad científica. No obstante, pueden asociarse a cada línea criopreservada en EMMA documentos que protejan la propiedad intelectual de los ratones depositados y puede solicitarse un periodo de hasta 2 años durante los cuales la línea está depositada en EMMA pero no aparece todavía en las bases de datos.
El nodo EMMA incluye la utilización de aisladores de jaulas de ratón, para permitir la recepción segura de ratones cuyo informe sanitario sea distinto al de los ratones alojados en el animalario del CNB. Se pretende con ello universalizar la criopreservación de líneas de ratón y ponerla a disposición de todos los investigadores interesados, con independencia de las características del animalario de origen.
COOKIES POLICY
A cookie is a text file that is stored on your computer or mobile device via a web server and only that server will be able to retrieve or read the contents of the cookie and allow the Web site remember browser preferences and navigate efficiently. Cookies make the interaction between the user and the website faster and easier.
General information
This Website uses cookies. Cookies are small text files generated by the web pages you visit, which contain the session data that can be useful later in the website. In this way this Web remembers information about your visit, which can facilitate your next visit and make the website more useful.
How do cookies?
Cookies can only store text, usually always anonymous and encrypted. No personal information is ever stored in a cookie, or can be associated with identified or identifiable person.
The data allow this website to keep your information between the pages, and also to discuss how to interact with the website. Cookies are safe because they can only store information that is put there by the browser, which is information the user entered in the browser or included in the page request. You can not run the code and can not be used to access your computer. If a website encrypts cookie data, only the website can read the information.
What types of cookies used?
The cookies used by this website can be distinguished by the following criteria:
1. Types of cookies as the entity that manages:
Depending on who the entity operating the computer or domain where cookies are sent and treat the data obtained, we can distinguish:
- Own cookies: are those that are sent to the user's terminal equipment from a computer or domain managed by the editor itself and from which provides the service requested by the user.
- Third party cookies: these are those that are sent to the user's terminal equipment from a machine or domain that is not managed by the publisher, but by another entity data is obtained through cookies.
In the event that the cookies are installed from a computer or domain managed by the editor itself but the information collected by these is managed by a third party can not be considered as party cookies.
2. Types of cookies as the length of time that remain active:
Depending on the length of time that remain active in the terminal equipment can be distinguished:
- Session cookies: cookies are a type designed to collect and store data while the user accesses a web page. Are usually used to store information that only worth preserving for the service requested by the user at any one time (eg a list of products purchased).
- Persistent cookies: cookies are a type of data which are stored in the terminal and can be accessed and treated for a period defined by the head of the cookie, and can range from a few minutes to several years.
3. Cookies types according to their purpose:
Depending on the purpose for which the data are processed through cookies, we can distinguish between:
- Technical cookies: these are those that allow the user to navigate through a web page or application platform and the use of different options or services it exist as, for example, control traffic and data communication, identify the session, access to restricted access parts, remember the elements of an order, make the buying process an order, make an application for registration or participation in an event, use security features while browsing store content for dissemination videos or sound or share content via social networks.
- Customization cookies: these are those that allow the user to access the service with some general characteristics based on a predefined set of criteria in the user terminal would eg language, the type of browser through which you access the service, the locale from which you access the service, etc.
- Analysis cookies: they are those that allow the responsible for them, monitoring and analyzing the behavior of users of the web sites that are linked. The information gathered through such cookies are used in measuring the activity of web sites, application or platform and for the profiling of user navigation of such sites, applications and platforms, in order to make improvements function data analysis how users use the service.
Management tool cookies
This Website uses Google Analytics.
Google Analytics is a free tool from Google that primarily allows website owners know how users interact with your website. Also, enable cookies in the domain of the site in which you are and uses a set of cookies called "__utma" and "__utmz" to collect information anonymously and reporting of website trends without identifying individual users..
For statistics of use of this website use cookies in order to know the level of recurrence of our visitors and more interesting content. This way we can concentrate our efforts on improving the most visited areas and make the user more easily find what they are looking for. On this site you can use the information from your visit for statistical evaluations and calculations anonymous data and to ensure the continuity of service or to make improvements to their websites. For more details, see the link below privacy policy [http://www.google.com/intl/en/policies/privacy/]
How to manage cookies on your computer: disabling and deleting cookies
All Internet browsers allow you to limit the behavior of a cookie or disable cookies within settings or browser settings. The steps for doing so are different for each browser, you can find instructions in the help menu of your browser.
If you decline the use of cookies, since it is possible thanks to the preferences menu of your browser or settings, reject, this website will continue to function properly without the use of the same.
Can you allow, block or delete cookies installed on your computer by setting your browser options installed on your computer:
- For more information about Internet Explorer click here.
- For more information on Chrome click here.
- For more information about Safari click here.
- For more information about Firefox click here.
Through your browser, you can also view the cookies that are on your computer, and delete them as you see fit. Cookies are text files, you can open and read the contents. The data within them is almost always encrypted with a numeric key corresponding to an Internet session so often has no meaning beyond the website who wrote it.
Informed consent
The use of this website on the other hand, implies that you paid your specific consent to the use of cookies, on the terms and conditions provided in this Cookies Policy, without prejudice to the measures of deactivation and removal of cookies that you can take, and mentioned in the previous section.







