
Genome editing: A new tool for generating genetically modified animal models
El 27 de mayo de 2014 se celebra en el Centro Nacional de Biotecnología del CSIC una jornada sobre edición genómica.
Organizada en por el Servicio de Transgénesis CNB-CBMSO y Sigma, quien patrocina el evento, se llevará a cabo en el Salón de Actos del CNB desde las 10:20 hasta las 14:30. La inscripción se realiza a través del formulario.
Programa:
- Silvia Di Meglio (Sigma-Aldrich, EMEA)
Using CRISPr and ZFN Technologies for creation of Transgenic animals - Belen Pintado (CNB-CBMSO, Madrid)
Genome editing. Impact on animal use - Roger Badia (Hospital Germans Trias i Pujol)
Working with ZFN: Practical issues learnt from HIV research - Ignacio Anegon (INSERM UMR, Nantes)
Gene editing of the rat genome using ZFNs and other gene-specific nucleases - Davide
Seruggia (CNB-CSIC, Madrid)
From TALENs to CRISPR: new tools for genome editing

La proteína de la envoltura del coronavirus del SARS determina su virulencia
En un estudio que investigadores del Centro Nacional de Biotecnología del CSIC (CNB) han publicado en la revista PLoS Pathogens se identifica la proteína viral clave para que el coronavirus del SARS produzca lesiones pulmonares.
 Luis Enjuanes, el investigador del CNB responsable principal de este trabajo, explica que “el coronavirus del SARS induce una respuesta inflamatoria incontrolada que causa daño pulmonar, edema, hipoxemia severa y eventualmente la muerte. Lo que hemos encontrado es que la actividad de la proteína E como transportadora de iones promueve las lesiones que causa el SARS”. Estos descubrimientos podrían llegar a usarse para desarrollar una terapia contra esta enfermedad.
Luis Enjuanes, el investigador del CNB responsable principal de este trabajo, explica que “el coronavirus del SARS induce una respuesta inflamatoria incontrolada que causa daño pulmonar, edema, hipoxemia severa y eventualmente la muerte. Lo que hemos encontrado es que la actividad de la proteína E como transportadora de iones promueve las lesiones que causa el SARS”. Estos descubrimientos podrían llegar a usarse para desarrollar una terapia contra esta enfermedad.
En colaboración con científicos de la Universitat Jaume I de Valencia, el investigador del CNB José Luis Nieto Torres ha generado coronavirus del SARS en los que su proteína de la envoltura carece de sus propiedad como transportadora de iones. Sin esta función, el virus se atenúa, reduciéndose la respuesta inflamatoria dañina que provoca a los ratones infectados.
Como hay varios virus muy patógenos con proteínas transportadoras de iones similares a las de estos coronavirus, Enjuanes comenta que sus resultados podrían ser relevantes en el estudio de infecciones de virus como el de la gripe A, el sida o la hepatitis C.
- Nieto-Torres JL, Dediego ML, Verdiá-Báguena C, Jimenez-Guardeño JM, Regla-Nava JA, Fernandez-Delgado R, Castaño-Rodriguez C, Alcaraz A, Torres J, Aguilella VM, Enjuanes L. Severe Acute Respiratory Syndrome Coronavirus envelope protein ion channel activity promotes virus fitness and pathogenesis. PLoS Pathog. 2014; 10(5): e1004077.
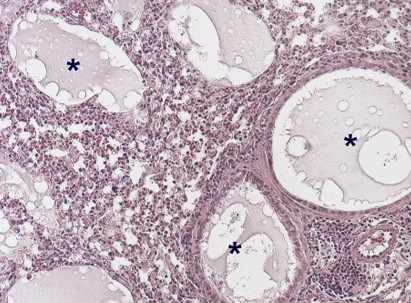
Descubren que la proteína Vps4 protege al genoma viral
Los virus ARN, entre los que se encuentran muchos patógenos para humanos, animales y plantas, replican sus genomas en membranas del interior de las células. En estas membranas los virus reclutan factores celulares que participan en el ensamblaje y actividades de los complejos replicativos. Estos agregados macromoleculares, que suelen ensamblarse en vesículas con aperturas al citosol o “esférulas”, se encargan de fabricar múltiples copias del genoma viral que se incorporarán posteriormente en las nuevas partículas virales infectivas. Aunque se han identificado numerosos factores necesarios para el ensamblaje funcional de los orgánulos de replicación viral se desconoce la función exacta de la mayoría de ellos.
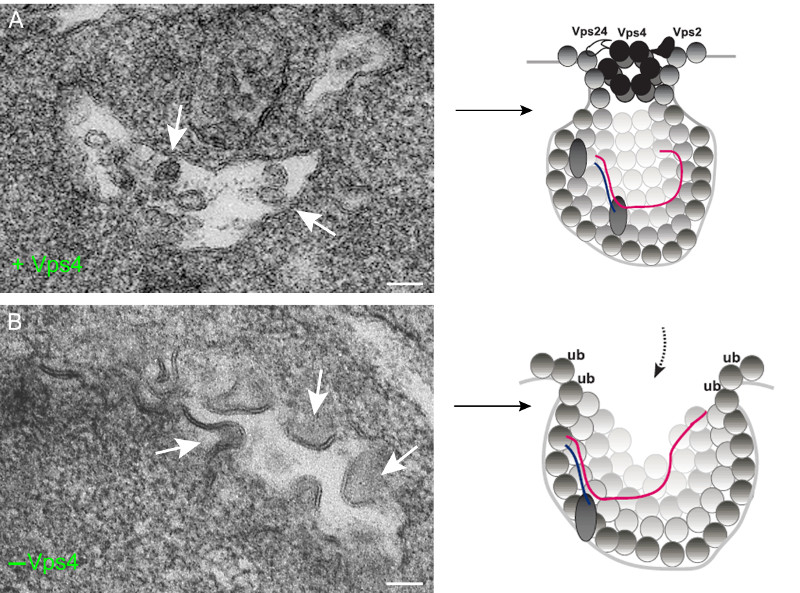 En colaboración con investigadores de la Universidad de Kentucky en los Estados Unidos, el laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Cristina Risco han encontrado evidencias de una función inesperada y sorprendente que desempeña la proteína celular Vps4 en la replicación de un virus ARN perteneciente a la familia de los Tombusvirus, patógenos que infectan plantas y que causan importantes pérdidas en cosechas.
En colaboración con investigadores de la Universidad de Kentucky en los Estados Unidos, el laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Cristina Risco han encontrado evidencias de una función inesperada y sorprendente que desempeña la proteína celular Vps4 en la replicación de un virus ARN perteneciente a la familia de los Tombusvirus, patógenos que infectan plantas y que causan importantes pérdidas en cosechas.
Vps4 es un componente de las proteínas celulares ESCRT y de la familia de las ATPasas AAA+ que usan ATP para remodelar estructuras macromoleculares en las células. Vps4 participa en una variedad de procesos biológicos como por ejemplo la fusión de membranas para la formación de vesículas. Utilizando nuevas técnicas de imagen en microscopía electrónica y según acaban de publicar en la revista PLoS Pathogens, los autores han demostrado que Vps4 participa en la estabilización del poro o cuello de las esférulas a través del cual se produce el intercambio de materiales con el citosol. Esta apertura controlada es necesaria para que el complejo replicativo desempeñe sus funciones y para que el ARN viral quede protegido de la degradación por nucleasas.
A diferencia de lo que ocurre en los procesos celulares habituales en los que participa, Vps4 se incorpora de manera estable en las vesículas virales o esférulas y pasa a formar parte del complejo replicativo. De hecho cuando Vps4 no está presente se forman esférulas totalmente abiertas, sin constricción o cuello y en las que el ARN viral de nueva síntesis está desprotegido. Los autores proponen que Vps4 y otras proteínas ESCRT son indispensables para la deformación de las membranas y el ensamblaje de los complejos replicativos de Tombusvirus. Es muy probable que otros virus de plantas y animales que transforman membranas celulares para construir esférulas también utilicen estas proteínas para ensamblar sus replicasas y proteger sus genomas recién sintetizados.
- Barajas D, Martín IFdC, Pogany J, Risco C, Nagy PD. Noncanonical role for the host Vps4 AAA+ ATPase ESCRT protein in the formation of Tomato bushy stunt virus replicase. PLoS Pathog. 2014; 10(4): e1004087.
Nueva estrategia para proteger a los linfocitos de la infección por HIV
Para que el HIV-1 entre en los glóbulos blancos, debe unirse a la proteína CD4 y a los correceptores CXCR4 o CCR5. Las cepas X4 del HIV-1 usan el correceptor CXCR4, mientras que las cepas R5 utilizan el CCR5. En personas recién infectadas por el virus del sida se aísla principalmente la cepa R5 del HIV-1, la cual infecta a células del sistema inmune tales como macrófagos, células dendríticas y linfocitos T CD4+. Estas células, que expresan CCR5 y algo de CXCR4, se encuentran en la mucosa genital y rectal y en los órganos linfoides del aparato digestivo. En la mitad de los pacientes, las cepas R5 mutan a cepas X4, un cambio que se asocia a la pérdida de linfocitos T CD4+ y al desarrollo de la enfermedad.
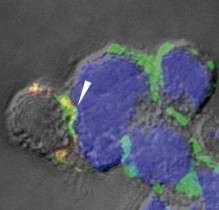 En un estudio que investigadores del Centro Nacional de Biotecnología del CSIC (CNB) publican en la revista Proceedings of the National Academy of Sciences USA se determina que CD4, CXCR4 y CCR5 forman oligómeros en las membranas de las células. Los resultados muestran además que la proporción de CXCR4 respecto a CCR5 controla la unión a las membranas de las cepas X4 del HIV-1, un paso necesario para que se produzca la entrada del HIV a las células hospedadoras. Los autores de este trabajo concluyen que según sean los niveles de CCR5 en las células diana, la unión de los virus X4 HIV-1 y por lo tanto su entrada en las células y la infección resultan bloqueadas. Este descubrimiento podría aplicarse para proteger a los linfocitos de la infección por HIV.
En un estudio que investigadores del Centro Nacional de Biotecnología del CSIC (CNB) publican en la revista Proceedings of the National Academy of Sciences USA se determina que CD4, CXCR4 y CCR5 forman oligómeros en las membranas de las células. Los resultados muestran además que la proporción de CXCR4 respecto a CCR5 controla la unión a las membranas de las cepas X4 del HIV-1, un paso necesario para que se produzca la entrada del HIV a las células hospedadoras. Los autores de este trabajo concluyen que según sean los niveles de CCR5 en las células diana, la unión de los virus X4 HIV-1 y por lo tanto su entrada en las células y la infección resultan bloqueadas. Este descubrimiento podría aplicarse para proteger a los linfocitos de la infección por HIV.
Sin la existencia en la actualidad de una vacuna efectiva contra el sida, “una nueva estrategia para impedir la infección del sida podría ser el diseño de fármacos que mimetizaran los efectos de CCR5 en las membranas celulares”, explica Mario Mellado, científico del CNB e investigador principal de este estudio. Comenta además que estos resultados ayudan a comprender por qué las cepas X4 del HIV-1 “conducen a una progresión más rápida de la enfermedad con un empeoramiento de los síntomas” y cómo la proporción de los receptores influye en la susceptibilidad a la infección.
- • Martínez-Muñoz L, Barroso R, Dyrhaug SY, Navarro G, Lucas P, Soriano SF, Vega, B, Costas C, Muñoz-Fernández MA, Santiago C, Rodríguez-Frade JM, Franco R, Mellado M. CCR5/CD4/CXCR4 oligomerization prevents HIV-1 gp120IIIB binding to the cell surface. Proc Natl Acad Sci USA. 2014; doi: 10.1073/pnas.1322887111.
2nd CNB Course on Introduction to Research
The Centro Nacional de Biotecnología (CNB-CSIC) is organising its 2nd Course on Introduction to Research for students in the final years of any university degree in science. The course is designed for qualified, highly motivated students who would like to contact the scientists at the CNB. The goal of this course is to provide an overview of the centre’s cutting-edge research facilities.
 The four-week course will be held from June 30 to July 25, 2014. Up to 12 selected students will be registered free of charge.
The four-week course will be held from June 30 to July 25, 2014. Up to 12 selected students will be registered free of charge.
For four weeks, the students will participate in the scientific activities of two research departments of their choice (2 weeks each), attend seminars on hot topics in today’s life sciences, and meet and talk with CNB scientists. Candidates are counselled to visit our website to learn about our projects and interests.
The completed application (doc/odt/pdf) form must be received before 30 May 2014, by e-mail to Alfonso Mora or by post to the Centro Nacional de Biotecnología (2nd Course on Introduction to Research), c/ Darwin #3, 28049 Madrid, Spain.
SELECTED STUDENTS
- Ricardo Sánchez de la Nieta Moreno
- Sara Herrera de la Mata
- Alberto Jesús González Hernández
- Andrés Sanz Morejón
- Iris Barragán Sánchez
- Dorota Kuczek
- Sergio Gómez López
- Isabel Tundidor Pérez
- Adolfo Alsina López
- Lorena Blázquez Conchillo
- Pilar Montero Calle
- Julio Téllez de Pablos
Cómo los potyvirus infectan las plantas sin matarlas
A lo largo de su evolución, los virus ARN han incorporado una serie de mecanismos para solventar las limitaciones de tener un genoma pequeño y relativamente sencillo. Deben asegurar que dentro de la célula hospedadora esté disponible tanto en el espacio como en el tiempo todo aquello necesario para su replicación, ensamblaje y amplificación. En este tipo de virus, una de las estrategias más comunes para disponer en un momento preciso de las proteínas necesarias es la proteolisis de una poliproteína.
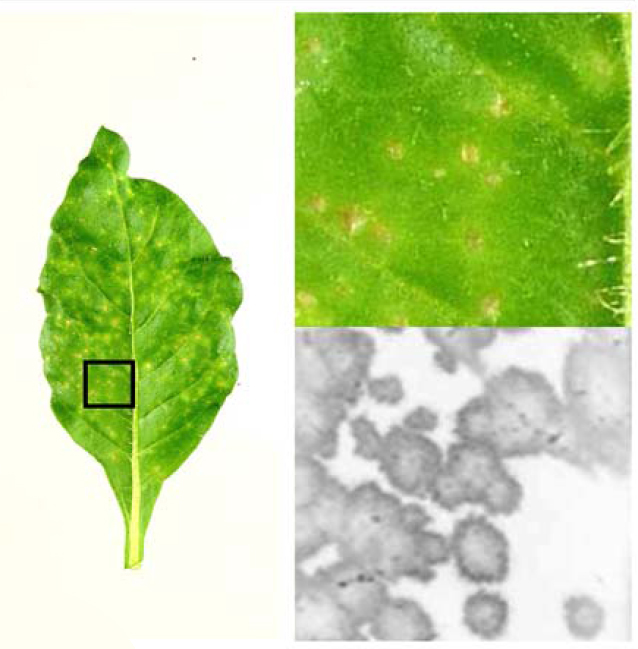 El grupo de investigación de Juan Antonio García y Carmen Simón centra sus esfuerzos en el Centro Nacional de Biotecnología del CSIC (CNB) en la endopeptidasa P1 de uno de los principales grupos de virus ARN que infectan a las plantas, los potyvirus. Aunque no se sabía su contribución exacta a la infección viral, algunos datos indicaban que P1 estaba implicada en la selección de hospedadores por parte del virus. Ahora, este grupo acaba de mostrar que el extremo amino-terminal de la proteína P1 de los potyvirus reduce la actividad autoproteolítica de esta enzima según el hospedador en el que se encuentre. Fabio Pasin, que realiza sus tesis doctoral en el CNB gracias a una beca de la Fundación La Caixa, ha observado que al eliminar esta región de la proteína P1 se acelera la replicación temprana del virus y aumenta la severidad de los síntomas. Todo ello, acompañado en la planta de un mayor número de marcadores marcadores de resitencia y una menor acumulación de virus.
El grupo de investigación de Juan Antonio García y Carmen Simón centra sus esfuerzos en el Centro Nacional de Biotecnología del CSIC (CNB) en la endopeptidasa P1 de uno de los principales grupos de virus ARN que infectan a las plantas, los potyvirus. Aunque no se sabía su contribución exacta a la infección viral, algunos datos indicaban que P1 estaba implicada en la selección de hospedadores por parte del virus. Ahora, este grupo acaba de mostrar que el extremo amino-terminal de la proteína P1 de los potyvirus reduce la actividad autoproteolítica de esta enzima según el hospedador en el que se encuentre. Fabio Pasin, que realiza sus tesis doctoral en el CNB gracias a una beca de la Fundación La Caixa, ha observado que al eliminar esta región de la proteína P1 se acelera la replicación temprana del virus y aumenta la severidad de los síntomas. Todo ello, acompañado en la planta de un mayor número de marcadores marcadores de resitencia y una menor acumulación de virus.
Estos resultados sugieren que el ajuste fino de la actividad de la proteasa P1 ha evolucionado para mantener la amplificación del virus a unos niveles no que causen demasiado daño al hospedador asegurando, eso si, la óptima replicación a largo plazo del virus. El estudio, que acaba de publicarse en la revista PLOS Pathogens, aporta nuevos datos experimentales que apoyan la teoría de que los virus mantienen unos niveles de replicación adecuados en dos hospedadores diferentes.
- Pasin F, Simón-Mateo C, García JA. The hypervariable amino-terminus of P1 protease modulates potyviral replication and host defense responses. PLoS Pathog. 2014; 10(3): e1003985.
Una pequeña proteína que regula el estrés hídrico
Las células eucariotas cuentan con el llamado sistema ubiquitino-proteosómico para degradar sus proteínas. Pero no es solo un sistema de deshecho, si no que al actuar de forma selectiva sobre algunas de ellas, es un mecanismo de control de la homesotasis de las proteínas, algo esencial en procesos vitales de las plantas como puedan ser la embriogénesis o la floración.
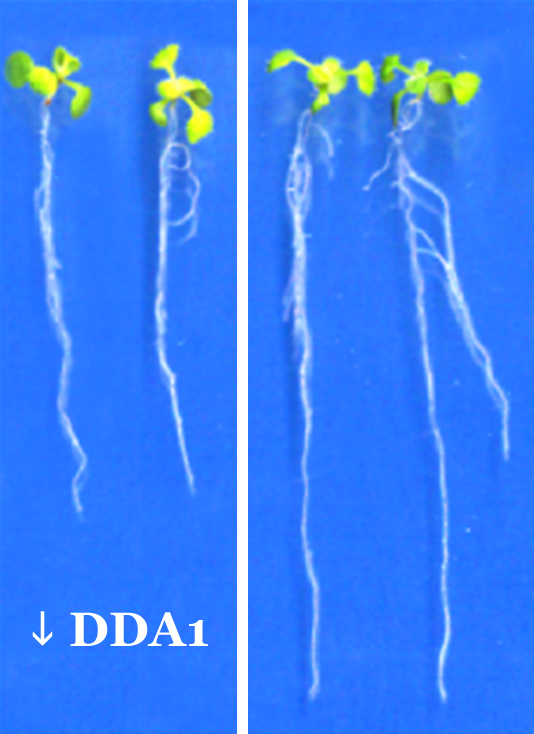 En su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo de Vicente Rubio estudia cómo el sistema ubiquitino-proteosómico regula el crecimiento de las plantas cuando están sometidas a estreses ambientales. Y acaban de descubrir en Arabidopsis que la pequeña proteína DDA1, existente tanto en plantas como en animales, es esencial para la estabilidad de los receptores de la hormona ácido abscísico (ABA). Esta hormona vegetal participa en la germinación de las semillas, controla el desarrollo de las plántulas e inicia las respuestas ante los estreses ambientales. Conocido como la "hormona de la sequía", el ácido abscísico es fundamental para que las plantas se adapten a la salinidad y al estrés osmótico, permitiéndolas vivir en condiciones adversas.
En su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo de Vicente Rubio estudia cómo el sistema ubiquitino-proteosómico regula el crecimiento de las plantas cuando están sometidas a estreses ambientales. Y acaban de descubrir en Arabidopsis que la pequeña proteína DDA1, existente tanto en plantas como en animales, es esencial para la estabilidad de los receptores de la hormona ácido abscísico (ABA). Esta hormona vegetal participa en la germinación de las semillas, controla el desarrollo de las plántulas e inicia las respuestas ante los estreses ambientales. Conocido como la "hormona de la sequía", el ácido abscísico es fundamental para que las plantas se adapten a la salinidad y al estrés osmótico, permitiéndolas vivir en condiciones adversas.
En su último trabajo, que acaban de publicar en la revista Plant Cell, María Luisa Irigoyen y Elisa Iniesto, junto a otros miembrops del grupo de Rubio, demuestran en la planta Arabidopsis thaliana que DDA1 forma parte de un complejo de proteínas implicado en la degradación proteosomal. Han encontrado que en las semillas y en las plántulas, esta pequeña proteínas se una alos recpetores del ácido abscísico facilitando así su degradación. De este modo, explica Rubio, tiene una función "negativa en la regulación de las respuestas a los estreses abióticos medidadas por ABA".
Los niveles de ABA son elevados en las semillas secas, lo que impide sus germinación. En las plantas adultas, estos niveles elevados lo que hacen es inhibir el crecimiento de las raíces y promover en las hojas el cierre de los estomas por los que la planta intercambia los gases. Cuando las condiciones ambientales son óptimas, los niveles de ABA disminuyen, los complejos que contiene DDA1 se activan más, degradando los receptores de ABA. Esto último consigue mantener los niveles necesarios de ABA a la vez que minimiza sus efectos inhibitorios (cierre de los estomas y retraso del crecimeinto) justo en el momento que no es necesario, durante la germinación o cuando no hay estrés.
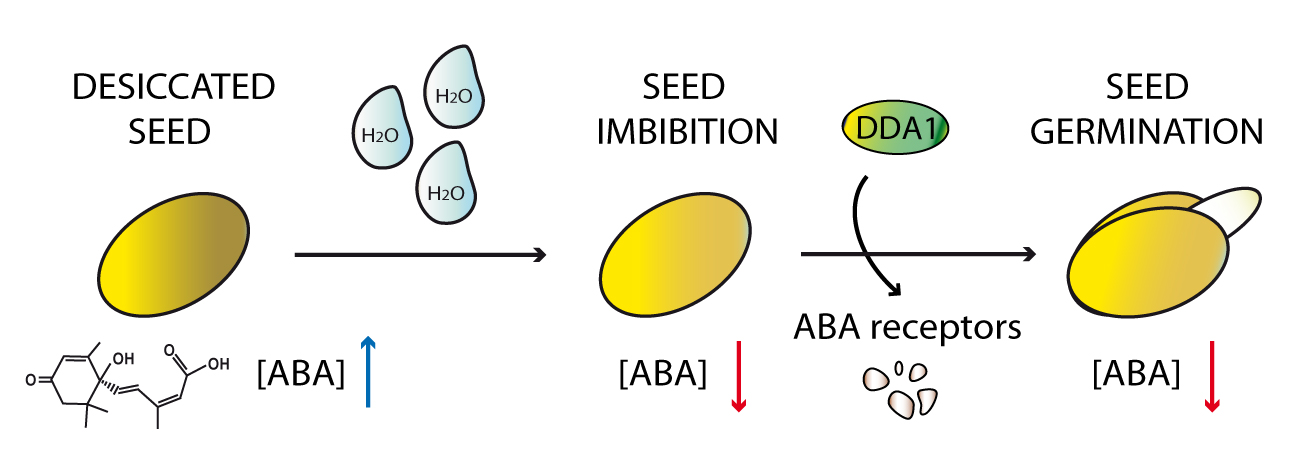
- Irigoyen ML, Iniesto E, Rodríguez L, Puga MI, Yanagawa Y, Pick E, Strickland E, Paz-Ares J, Wei N, De Jaeger G, Rodriguez PL, Deng XW, Rubio V. Targeted degradation of abscisic acid receptors is mediated by the ubiquitin ligase substrate adaptor DDA1 in Arabidopsis. Plant Cell. 2014; doi: 10.1105/tpc.113.122234.
Descubren un mecanismo que regula el tamaño de los cilios
En la superficie de muchos tipos de células los mamíferos podemos encontrar cilios, los cuales participan en multitud de procesos fisiológicos que van desde el crecimiento celular y el desarrollo hasta la percepción del ambiente en el que se encuentran. Su importancia se pone de manifiesto si tenemos en cuenta algunas enfermedades causadas por defectos en estos orgánulos: el hidrocéfalo, la anosmia (pérdida del olfato) o la retinitis pigmentaria.
 El grupo de investigación del Centro Nacional de Biotecnología del CSIC dirigido por Karel H.M. van Wely estudia las proteínas involucradas en la formación y en el desensamblaje de los cilios. Si bien se conoce desde hace tiempo su regulación asociada a la mitosis de la céllulas, en este laboratorio del CNB se acaban de encontrar un mecanismo que regula el tamaño de los cilios cuando la célula no se está dividiendo. Los experimentos que ha realizado en este laboratorio Ainhoa Sánchez de Diego, y que acaba de publicar en la revista Nature Communications, muestran que cuando las céllulas no están creciendo la distribución subcelular de la deacetilasa HDAC6 es esencial para controlar el tamaño de los cilios.
El grupo de investigación del Centro Nacional de Biotecnología del CSIC dirigido por Karel H.M. van Wely estudia las proteínas involucradas en la formación y en el desensamblaje de los cilios. Si bien se conoce desde hace tiempo su regulación asociada a la mitosis de la céllulas, en este laboratorio del CNB se acaban de encontrar un mecanismo que regula el tamaño de los cilios cuando la célula no se está dividiendo. Los experimentos que ha realizado en este laboratorio Ainhoa Sánchez de Diego, y que acaba de publicar en la revista Nature Communications, muestran que cuando las céllulas no están creciendo la distribución subcelular de la deacetilasa HDAC6 es esencial para controlar el tamaño de los cilios.
En esta publicación se demuestra que no sólo es necesario que una quinasa active a HDAC6, es también clave para actuar sobre un sustrato u otro la localización de la deacetilasas en el interior de la célula. La proteína Dido3, descubierta por este mismo grupo, es fundametal a la hora de regular tanto los niveles de HDCA6 en la base de los cilios como el grado de acetilación de la tubulina.
- Sánchez de Diego A, Guerrero AA, Martínez-A C, van Wely KHM. Dido3-dependent HDAC6 targeting controls cilium size. Nat Commun. 2014; doi: 10.1038/ncomms4500.
Nuevo papel de las p38 MAP quinasas en la artritis reumatoide
La artritis reumatoide es una enfermedad autoinmune que aunque afecta principalmente a las articulaciones, causa también inflamación crónica en multitud de tejidos y órganos. Se calcula que afecta al 1% de la población, con síntomas desde la hiperplasia sinovial hasta la destrucción de las articulaciones. Todos estos síntomas son consecuencia de una liberación continua y desregulada de citoquinas inflamatorias.
 El grupo de Ana Cuenda investiga en el Centro Nacional de Biotecnología del CSIC (CNB) el papel que juegan en la regulación de la producción de citoquinas las cuatro quinasas de la familia p38MAPK. En concreto, centran sus esfuerzos en ver la implicación específica de cada una de ellas en la patogénesis de la artritis reumatoide con la intención de que se puedan desarrollar nuevos tratamientos más eficaces y seguros que los actuales.
El grupo de Ana Cuenda investiga en el Centro Nacional de Biotecnología del CSIC (CNB) el papel que juegan en la regulación de la producción de citoquinas las cuatro quinasas de la familia p38MAPK. En concreto, centran sus esfuerzos en ver la implicación específica de cada una de ellas en la patogénesis de la artritis reumatoide con la intención de que se puedan desarrollar nuevos tratamientos más eficaces y seguros que los actuales.
En un trabajo que acaban de publicar en colaboración con investigadores del Hospital '12 de Octubre' de Madrid en la revista Arthritis and Rheumatism, el grupo de Cuenda presenta el descubrimiento de que la eliminación de los genes de las quinasas p38γ y p38δ “reduce significativamente tanto la incidencia como la severidad de la artritis“. Induciendo artritis en ratones con colágeno, la falta de estas dos proteínas se acompañaba además con una menor producción de citoquinas. Estos resultados, comenta Cuenda, muestran que las quinasas p38γ y p38δ son cruciales para regular la inflamación de las articulaciones y la destrucción de los huesos que se producen en la artrtitis y, por tanto, serían dos nuevas dianas terapéuticas en el tratamiento de la artritis reumatoide.
- Criado G, Risco A, Alsina-Beauchamp D, Pérez-Lorenzo MJ, Escós A, Cuenda A. Alternative p38 mitogen-activated protein kinases are essential for collagen-induced arthritis. Arthritis Rheum. doi: 10.1002/art.38327.
Nueva generación de la base de datos de proteínas del centrosoma
El gobierno de la Comunidad de Madrid recientemente financió el proyecto de investigación PROFUN II: Interactomics of the Centrosome (CS Interactomics) que tiene como objetivo el estudio de la interactómica del centrosoma, un orgánulo celular clave en el proceso de organización de la arquitectura celular. En este proyecto, varios grupos de investigadores de Madrid están uniendo esfuerzos para proporcionar un impulso cualitativo al conocimientos del centrosoma, una pieza fundamental para la comprensión de la célula.
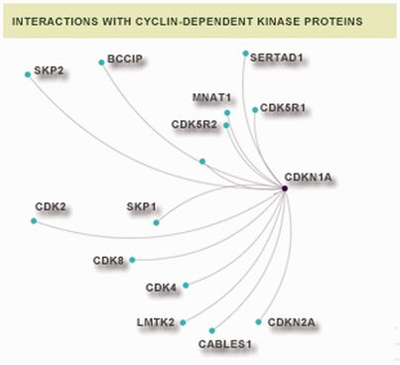 En este contexto, el grupo de Bioinformática funcional del CNB dirigido por Alberto Pascual-Montano ha desarrollado CentrosomeDB, una base de datos que recoge e integra información relevante sobre el centrosoma. Esta base de datos ofrece diferentes perspectivas para el estudio de las proteínas del centrosoma, incluyendo su estructura, su función y su evolución. Además, contiene información sobre las homologías con las proteínas de otras especies, así como datos de su función, su relación con enfermedades (SNP) o la estructura tridimensional de las mismas. Se han incorporado también, resultados sobre las interacciones demostradas entre las proteínas.
En este contexto, el grupo de Bioinformática funcional del CNB dirigido por Alberto Pascual-Montano ha desarrollado CentrosomeDB, una base de datos que recoge e integra información relevante sobre el centrosoma. Esta base de datos ofrece diferentes perspectivas para el estudio de las proteínas del centrosoma, incluyendo su estructura, su función y su evolución. Además, contiene información sobre las homologías con las proteínas de otras especies, así como datos de su función, su relación con enfermedades (SNP) o la estructura tridimensional de las mismas. Se han incorporado también, resultados sobre las interacciones demostradas entre las proteínas.
Desde su publicación, esta nueva jerramienta ha recibido ya más de 10.000 visitas al ser probablemente la base de datos más completa de proteínas del centrosoma. En estos momentos contiene 1.053 proteínas centrosomales humanas y 304 de Drosophila melanogaster, una mejora significativa respecto a otras herramientas y repositorios existentes.
La base de datos se puede consultar públicamente en http://centrosome.cnb.csic.es.
- Alves-Cruzeiro JM, Nogales-Cadenas R, Pascual-Montano AD. CentrosomeDB: a new generation of the centrosomal proteins database for Human and Drosophila melanogaster. Nucleic Acids Res. 2014; 42(1): D430-D436.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.





