Nueva edición de los Premios Fundación Biogen para Jóvenes Investigadores
Desvelan cómo ciertos patógenos regulan la expresión de genes de resistencia a antibióticos
IX Ciclo de Seminarios Júnior
XX Ciclo de Seminarios del CNB
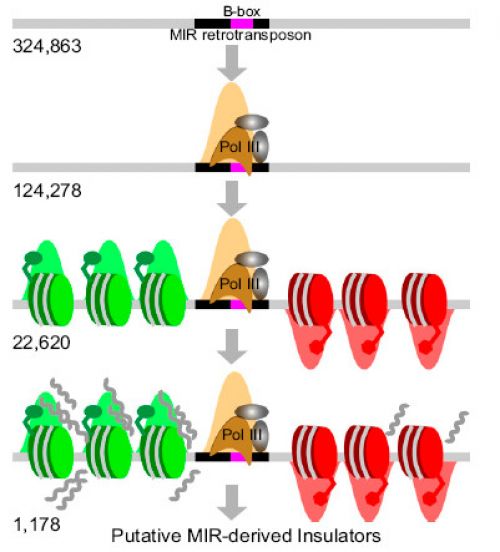
Las secuencias MIR actúan como elementos reguladores del genoma humano
Identifican el mecanismo con el que la planta de la patata controla la formación de ramas y estolones
La Sociedad Española de Virología premia a Pablo Gastaminza
El investigador del CNB Pablo Gastaminza ha recibido el premio Virólogo Joven 2015 de la Sociedad Española de Virología por su brillante carrera científica. Gastaminza fue galardonado durante la celebración en Madrid del XIII Congreso Nacional de Virología.
 Gastaminza se incorporó al Centro Nacional de Biotecnología en 2010. Su laboratorio estudia los procesos celulares y moleculares que subyacen en diferentes aspectos de la biología y patogénesis del virus de la hepatitis C (VHC) con la intención de descubrir nuevas estrategias de lucha contra el virus. Recientemente describieron un factor celular, el receptor sigma-1 (S1R), como potencial diana para terapia antiviral.
Gastaminza se incorporó al Centro Nacional de Biotecnología en 2010. Su laboratorio estudia los procesos celulares y moleculares que subyacen en diferentes aspectos de la biología y patogénesis del virus de la hepatitis C (VHC) con la intención de descubrir nuevas estrategias de lucha contra el virus. Recientemente describieron un factor celular, el receptor sigma-1 (S1R), como potencial diana para terapia antiviral.
Con el uso de un sistema de análisis desarrollado por ellos mismos, identificaron en su laboratorio 12 fármacos empleados en distintas enfermedades que selectivamente bloquean la entrada del virus de la hepatitis C en nuestras células. Sus artículos han sido citados en más de 2.500 ocasiones.
Encuentro internacional de científicos y comunicadores
Durante los días 7 y 8 de octubre, la isla de la Palma acogerá la primera edición del Congreso 100xCiencia (Communicating Frontier Science). En él, los 20 centros de investigación españoles reconocidos por el programa de excelencia “Severo Ochoa” (SO) acercaremos nuestra ciencia de frontera a los medios de comunicación y al público general. También será un foro de debate sobre la comunicación y divulgación de la ciencia: mediante mesas redondas y ponencias de comunicadores y periodistas internacionales, reflexionaremos sobre las estrategias de comunicación y el impacto de la ciencia en los medios y en la sociedad.
 100xCiencia pretende ser un lugar de encuentro entre científicos, periodistas y divulgadores. “Con este foro –explica Rafael Rebolo, presidente del Comité Organizador- queremos impulsar y crear nuevas formas de colaboración que nos permitan acercar más y mejor nuestras investigaciones a la sociedad en general y, en particular, a los más jóvenes. El futuro de una sociedad está muy ligado a la calidad de su ciencia, al trabajo y entrega de sus científicos, pero también al apoyo y los recursos que reciben. Incrementar el interés de la gente por la ciencia es esencial para lograr más apoyo. Por eso queremos y necesitamos transmitir mejor lo que hace la ciencia por todos”.
100xCiencia pretende ser un lugar de encuentro entre científicos, periodistas y divulgadores. “Con este foro –explica Rafael Rebolo, presidente del Comité Organizador- queremos impulsar y crear nuevas formas de colaboración que nos permitan acercar más y mejor nuestras investigaciones a la sociedad en general y, en particular, a los más jóvenes. El futuro de una sociedad está muy ligado a la calidad de su ciencia, al trabajo y entrega de sus científicos, pero también al apoyo y los recursos que reciben. Incrementar el interés de la gente por la ciencia es esencial para lograr más apoyo. Por eso queremos y necesitamos transmitir mejor lo que hace la ciencia por todos”.
Contaremos con la presencia de una delegación invitada de reconocidos comunicadores de la ciencia y representantes de grandes medios de comunicación nacionales e internacionales que tomarán parte activa durante la celebración del evento. El programa completo se irá actualizando en las próximas semanas. La inscripción ya está abierta.
Los centros “Severo Ochoa” cubrimos prácticamente todas las áreas del conocimiento, desde astrofísica y matemáticas hasta medio ambiente y biomedicina, pasando por las humanidades. El foro ofrecerá una visión completa de la investigación de frontera que se realiza en España, con ponencias divulgativas en las que representantes de cada centro presentarán a los periodistas los nuevos resultados de investigación, así como los retos de futuro en su área de acción.
En las mesas redondas debatiremos cuestiones de interés para comunicadores y científicos, como la presencia de la ciencia en los medios de comunicación, el papel de la investigación en los grandes retos de la sociedad actual, el impacto de las acciones de divulgación y la situación de política científica en España y Europa. El congreso se complementa con actividades de divulgación dirigidas a los habitantes de La Palma.
Curso 'La biotecnología ante los retos de la sociedad actual'
El Centro Nacional de Biotecnología (CNB) del CSIC, acreditado como centro de excelencia Severo Ochoa, invita a catedráticos y profesores de enseñanza secundaria a participar en la primera edición del curso presencial de actualización docente La biotecnología ante los retos de la sociedad actual que se impartirá del 29 de junio al 3 de julio de 2015, en el campus de Cantoblanco, Madrid.
El curso, equivalente a 2 créditos, ha sido autorizado por la Dirección General de Mejora de la Calidad de la Enseñanza, está dirigido a profesores de Biología, Geología, Química y Tecnología, y será impartido por investigadores del CNB con amplia experiencia en comunicación científica. Tiene como objetivo contribuir a actualizar la formación de los docentes en Microbiología, Virología, Inmunología, Oncología, Genética Molecular de Plantas y Biología Molecular, Estructural y Celular.
La inscripción estará abierta hasta el 12 de junio de 2015 y se realizará por correo electrónico a la dirección . Las plazas se adjudicarán en base al orden de recepción de las solicitudes.
La información del curso puede consultarse en el sitio web de la convocatoria:
La Fundación Lilly premia a José Luis Martínez
Durante la sesión de hoy del simposio internacional Microbiología: TRANSMISIÓN, José Luis Martínez recibirá el Premio Fundación Lilly a una Carrera Distinguida.
 Desde 2001, el grupo del Centro Nacional de Biotecnología del CSIC dirigido por Martínez estudia los mecanismos por los que las bacterias de origen medioambiental adquieren resistencias a los antibióticos. Sus resultados han sido esenciales para comprender cómo evolucionan las bacterias durantres las infecciones crónicas.
Desde 2001, el grupo del Centro Nacional de Biotecnología del CSIC dirigido por Martínez estudia los mecanismos por los que las bacterias de origen medioambiental adquieren resistencias a los antibióticos. Sus resultados han sido esenciales para comprender cómo evolucionan las bacterias durantres las infecciones crónicas.
Con su investigación analiza el papel que juegan los ecosistemas naturales en la adquisición y la evolución de la resistencia a los antibióticos. Aunque los antibióticos se empezaron a usar como medicamentos en los años 40, Martínez explica que la adquisición de resitencia a ellos por parte de las bacterias "es el resultado de millones de años de evolución en ecosistemas naturales no clínicos".
Su trabajo aporta información para la búsqueda de nuevos fármacos que puedan ayudar a reducir la resistencia a los antibióticos. Entre las dianas terapéuticas que ha encontrado, actualmente centra sus estudios en un sistema de bombeo múltiple de drogas presente en todos los seres vivos y que contribuye tanto a la resistencia a la quimioterapia como a los antibióticos. Su grupo encontró recientemenet que la expresión de la bomba SmeDEF de Stenotrophomonas maltophilia se iniciada en presencia del biocida triclosa, suguiriendo que los biocidas que usamos habitualmente podrían llegar a seleccionar microrganismos resistentes a los antibióticos.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.