
Dido controla la proliferación y diferenciación de las células madre
Las células "madre" embrionarias reciben este nombre por su capacidad de generar cualquier tipo celular de las tres capas del embrión a partir de las cuales se originarán los tejidos y órganos en el individuo adulto: el endodermo, el mesodermo o el ectodermo (diferenciación). Además, y al contrario que las células adultas especializadas, las células madre tienen la asombrosa capacidad de poder multiplicarse indefinidamente en el laboratorio (división), lo que las hace tremendamente útiles en el campo de la medicina regenerativa.
 En la actualidad, los científicos tratan de comprender los mecanismos moleculares que regulan en las células madre el equilibrio entre ambos procesos, la división y la diferenciación. Últimamente se han identificado genes que permiten reprogramar células especializadas y convertirlas en células madre pero aun hay muchos genes específicos de estas células cuya función es desconocida.
En la actualidad, los científicos tratan de comprender los mecanismos moleculares que regulan en las células madre el equilibrio entre ambos procesos, la división y la diferenciación. Últimamente se han identificado genes que permiten reprogramar células especializadas y convertirlas en células madre pero aun hay muchos genes específicos de estas células cuya función es desconocida.
Un ejemplo es el gen Dido al que dedica sus esfuerzos el grupo del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Carlos Martínez-A. Este gen aparece evolutivamente en vertebrados y se manifiesta en tres proteínas distintas: Dido1, Dido2 y Dido3. Esta última se expresa en células madre y se modula su expresión en las células especializadas. Se une a estructuras esenciales para la división celular como son el centrosoma y el complejo sinaptonémico. Alteraciones de esta proteína causan síndromes preleucémicos y tumorales y bloquean la capacidad de diferenciarse de las células madre en cualquier tipo celular específico (diferenciación) manteniendo su capacidad de división (proliferación).
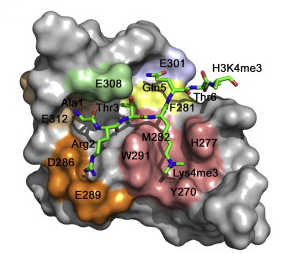
En colaboración con investigadores de la Universidad de Colorado, los científicos del CNB acaban de publicar un trabajo en el que identifican al dominio PH de Dido3, y determinan su estructura cristalina a 1.8 A de resolución, como el responsable de la unión al cromosoma. Esta unión causaría modificaciones en la expresión de diferentes genes que le dan a las células madre su capacidad de dividirse y diferenciarse en células adultas.
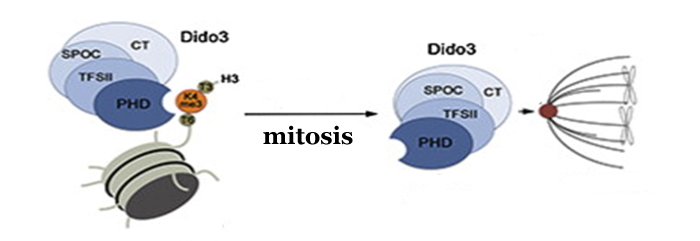
Además de identificar la zona de la proteína que se une a las histonas que organizan los cromosomas, los investigadores han profundizado en cómo se regula su función según el momento del ciclo celular en que se encuentre la célula. Sus datos indican que cuando las células madre se diferencian, Dido3 es desplazada del cromosoma por el aumento de Dido1. Esto conlleva una disminución en la expresión de genes que dotan a las células madre de su pluripotencia.
En este mismo aspecto, durante la mitosis han observado que Dido3 pasa de estar unido a la cromatina a desplazarse hasta el huso mitótico que dispone a los cromosomas en el ecuador de la célula como paso previo a separarlos hacia los polos cuando se va a producir la división de una célula en dos. De este modo, queda descrita por primera vez a través de Dido relación entre la expresión de genes durante el desarrollo embrionario y la regulación del ciclo celular
- Gatchalian J, Fütterer A, Rothbart SB, Tong Q, Rincon-Arano H, Sánchez de Diego A, Groudine M, Strahl BD, Martínez-A C, van Wely KH, Kutateladze TG. Dido3 PHD modulates cell differentiation and division. Cell Rep 2013 Jul 2. pii: S2211-1247(13)00292-1.
Identifican las primeras bases moleculares de la percepción de arsénico en plantas
La presencia de arsénico en la naturaleza debido a las erupciones volcánicas ha sido una de las amenazas más importantes para el desarrollo de la vida en la tierra. Hoy en día el arsénico presente en los suelos, puede ser solubilizado en las aguas subterráneas exponiendo a los organismos a uno de los carcinógenos más potentes que se conocen. De hecho la exposición a arsénico es responsable del envenenamiento masivo más importante que ha sufrido la humanidad en toda su historia.
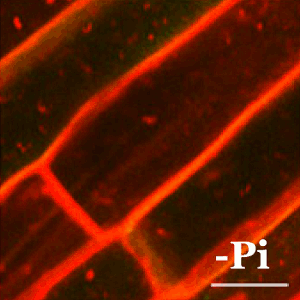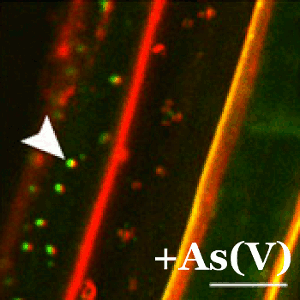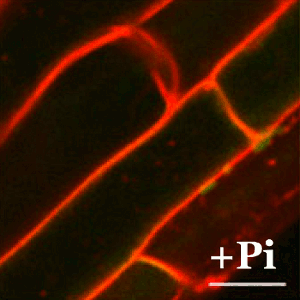 En plantas, la tolerancia al arsénico es esencial para su supervivencia ya que la forma química más abundante en la tierra (el arseniato) guarda una gran similitud con el fosfato y las plantas lo incorporan fácilmente a sus células a través de los transportadores de fosfato. En el CNB, el grupo dirigido por Antonio Leyva estudia las bases moleculares de los mecanismos de percepción de arsénico en plantas con el fin de identificar y desarrollar plantas que eviten la exposición de este carcinógeno a los seres vivos.
En plantas, la tolerancia al arsénico es esencial para su supervivencia ya que la forma química más abundante en la tierra (el arseniato) guarda una gran similitud con el fosfato y las plantas lo incorporan fácilmente a sus células a través de los transportadores de fosfato. En el CNB, el grupo dirigido por Antonio Leyva estudia las bases moleculares de los mecanismos de percepción de arsénico en plantas con el fin de identificar y desarrollar plantas que eviten la exposición de este carcinógeno a los seres vivos.
En este trabajo, merecedor de un comentario especial por parte de los editores de la revista Plant Cell, el grupo de Leyva ha descubierto que cuando las plantas detectan el arseniato impiden de forma inmediata su captación mediante la represión y deslocalización del transportador de fosfato. Este sistema está controlado mediante la acción de una proteína represora conocida como WRKY6.
En colaboración con otros dos grupos del CNB, han podido además descubrir que el arseniato provoca la activación masiva de transposones, unos elementos del ADN móviles capaces de “saltar” de un sitio a otro del genoma en respuesta a estrés y por tanto actuar como agentes mutagénicos. Una activación que también está controlada por el represor WRKY6.
Estos resultados indican que las plantas poseen un mecanismo de percepción del arseniato que controla la activación de transposones y la incorporación del arseniato. Todo ello proporciona un sistema integrado de tolerancia y de estabilidad genómica.
- Castrillo G, Sánchez-Bermejo E, de Lorenzo L, Crevillén P, Fraile-Escanciano A, Tc M, Mouriz A, Catarecha P, Sobrino-Plata J, Olsson S, Leo Del Puerto Y, Mateos I, Rojo E, Hernández LE, Jarillo JA, Piñeiro M, Paz-Ares J, Leyva A. WRKY6 transcription factor restricts arsenate uptake and transposon activation in Arabidopsis. Plant Cell 2013 Aug 6 tpc.113.114009.
IV Curso de Proteómica Cuantitativa
Organizado por el Servicio de Proteómica del Centro Nacional de Biotecnología del CSIC, del 7 al 11 de octubre de 2013 se va a celebrar en Madrid el IV Curso de Proteómica Cuantitativa.
 Este curso de Proteómica Cuantitativa pretende ofrecer una exhaustiva visión teórica y práctica de algunas las aproximaciones experimentales más utilizadas en proteómica diferencial. Como puede comprobarse en el programa del curso, a lo largo de cuatro días se analizarán en detalle técnicas como el marcaje isotópico diferencial con reactivos como ICPL (Isotope-coded protein labelling) y iTRAQ, seguido de fraccionamiento por cromatografía líquida y análisis mediante espectrometría de masas y técnicas de proteómica cuantitativa dirigida (MRM).
Este curso de Proteómica Cuantitativa pretende ofrecer una exhaustiva visión teórica y práctica de algunas las aproximaciones experimentales más utilizadas en proteómica diferencial. Como puede comprobarse en el programa del curso, a lo largo de cuatro días se analizarán en detalle técnicas como el marcaje isotópico diferencial con reactivos como ICPL (Isotope-coded protein labelling) y iTRAQ, seguido de fraccionamiento por cromatografía líquida y análisis mediante espectrometría de masas y técnicas de proteómica cuantitativa dirigida (MRM).
Las clases teóricas se combinarán con clases prácticas que permitirán al investigador familiarizarse con estas técnicas y descubrir todo su potencial La proteómica permite la identificación y caracterización de las proteínas presentes en proteomas y subproteomas complejos
La información más actualizada puede encontrarse en la página web del IV Curso de Proteómica Cuantitativa.
El Campus de Biociencias de Madrid Norte busca un director
El Campus de Biociencias de Madrid Norte es un proyecto del área de las Ciencias de la Vida y de la Salud del Campus de Excelencia UAM+CSIC formado por los principales agentes responsables de la I+D+ i en biociencias en el área norte de Madrid. El objetivo del Campus es crear un entorno de actividad científica a través de la colaboración entre sus agentes y empresas relacionadas para fomentar el desarrollo de colaboraciones tecnológicas en Ciencias de la Vida y la Salud.
 La principal función del Director/a será poner en marcha, dirigir y administrar el Campus. Será el responsable de negociar con los diferentes agentes involucrados y promover colaboraciones nacionales e internacionales en línea con la estrategia del Campus de Excelencia.
La principal función del Director/a será poner en marcha, dirigir y administrar el Campus. Será el responsable de negociar con los diferentes agentes involucrados y promover colaboraciones nacionales e internacionales en línea con la estrategia del Campus de Excelencia.
Requisitos:
- Licenciado/a o doctor/a. Se valorará la formación en Ciencias de la Vida.
- Experiencia demostrable en gestión, promoción, evaluación y coordinación de actividades de I+D+i en ciencias y tecnología y en transferencia de resultados. Se valorará experiencia científica.
- Experiencia en planificación y gestión estratégica de Centros de Excelencia públicos o privados, fundaciones de investigación, institutos de saludo pública o entidades públicas.
- Liderazgo y visión estratégica.
- Conocimiento del entorno científico empresarial, especialmente en el ámbito de la salud. Se valorará positivamente experiencia en el sector de la biotecnología y del sector de la salud en España, y en Madrid, en particular.
- Conocimiento de las políticas nacionales e internacionales en biociencias y en I+D+ i.
- Conocimiento de la red nacional e internacional de instituciones públicas y privadas en biociencias
- Inglés y Español fluidos.
Se precisa incorporación inmediata, dedicación completa y disponibilidad para viajar.
Modalidad contractual: contrato por obra o servicio.
Jornada: completa.
Retribución: a convenir en función de la valía el candidato seleccionado.
La selección de los candidatos se realizará mediante la valoración del curriculum, entrevista personal y la valoración de la memoria estratégica que se solicitará en la primera entrevista.
Fecha límite para entrega de candidaturas: jueves 31 de julio hasta las 14.00 horas.
Dirigir las solicitudes por correo electrónico a o por correo postal a la FUAM, Calle Einstein, nº 13, planta 2ª. 28049 MADRID.
Indicar referencia: DCBNM.
*El puesto podrá declararse desierto si la FUAM considera que ninguno de los candidatos cumple los requisitos.
Describen cómo el interferón protege contra los hongos
Para luchar contra las infecciones, nuestro organismo cuenta con un tipo de moléculas conocidas como interferones que consiguen bloquear la multiplicación de los virus y activar a los glóbulos blancos que atacan tanto a estos como a las bacterias. En su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo dirigido por Carlos Ardavín estudia el funcionamiento de unas células inmunitarias que producen interferón, las células dendríticas.
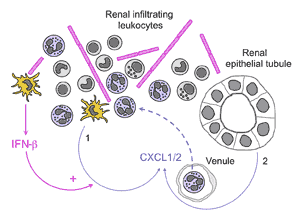 A parte del efecto contra virus y bacterias, hace años se descubrió que “las células dendríticas son clave en la inmunidad contra el hongo Candida albicans” explica Ardavín, quien acaba de publicar en la revista Immunity que para que puedan actuar contra dicho hongo es necesario que las células dendríticas produzcan interferón de tipo I (α y β). Sólo así, las células dendríticas reducen la mortalidad en infecciones causadas por Candida albicans en ratones.
A parte del efecto contra virus y bacterias, hace años se descubrió que “las células dendríticas son clave en la inmunidad contra el hongo Candida albicans” explica Ardavín, quien acaba de publicar en la revista Immunity que para que puedan actuar contra dicho hongo es necesario que las células dendríticas produzcan interferón de tipo I (α y β). Sólo así, las células dendríticas reducen la mortalidad en infecciones causadas por Candida albicans en ratones.
Profundizando en el mecanismo molecular, los científicos del CNB han descubierto el papel esencial de dos proteínas llamadas Dectin-1 y Dectin-2 para que las células dendríticas produzcan Interferón β cuando el organismo sufre una infección por dicho hongo. Sin ellas, no se produce el interferón y, entre otras cosas, los neutrófilos encargados de luchar contra la infección no se desplazan hasta el lugar donde se encuentra C. albicans.
- Del Fresno C, Soulat D, Roth S, Blazek K, Udalova I, Sancho D, Ruland J, Ardavín C. Interferon-β production via Dectin-1-Syk-IRF5 signaling in Dendritic Cells is crucial for immunity to C. albicans. Immunity 2013 Jun 27;38(6):1176-86.
Describen cómo la proteína AddAB recorre el ADN para su reparación
Produciéndose cerca de 10.000 alteraciones diarias en el genoma de cada célula, el trabajo que hacen proteínas reparadoras del ADN como las que estudia en su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) el grupo dirigido por el biofísico Fernando Moreno-Herrero es tremendamente importante.
 Junto a bioquímicos de la Universidad de Bristol, han publicado en la revista PNAS el mecanismo de exploración o escaneo que utiliza la proteína AddAB en el proceso de reparación de un corte de la doble hélice de ADN. Como nos explica Moreno-Herrero, reparar los daños en esencial ya que su acumulación puede acabar provocando "la muerte celular o incluso degenerar en una célula cancerígena".
Junto a bioquímicos de la Universidad de Bristol, han publicado en la revista PNAS el mecanismo de exploración o escaneo que utiliza la proteína AddAB en el proceso de reparación de un corte de la doble hélice de ADN. Como nos explica Moreno-Herrero, reparar los daños en esencial ya que su acumulación puede acabar provocando "la muerte celular o incluso degenerar en una célula cancerígena".
Con anterioridad, el grupo del CNB había demostrado que para que la proteína AddAB recorriera el cromosoma separando las dos hebras necesita estar unida a unas secuencias del genoma llamadas Chi. Ahora además, utilizando la técnica de pinzas magnéticas, han sido capaces de estudiar molécula a molécula cómo recorre el ADN, a qué velocidad y hasta las pausas que hacen. Sus resultados muestran que las características de la translocación a lo largo del ADN dependen del contenido en citosina y guanina y de la presencia de las secuencias Chi.
Desde el CNB, la investigadora postdoctoral Carolina Carrasco nos explica que en su estudio han podido ver "que cuando la proteína encuentra una verdadera secuencia Chi, realiza una pausa en esta posición después de la cual reinicia nuevamente su trayectoria". Y es que cuando AddAB se encuentra con secuencias parecidas a Chi, también se detiene en un intento fallido de reconocer estas pseudo-Chi, siendo la duración de las pausas en estos casos menores.
Basándose en la capacidad de dicha proteína de reconocer específicamente las secuencias Chi, los investigadores han propuesto un modelo mecánico para explicar el origen de ambas pausas. La forma de la pausa y el aparente cambio de velocidad después de la misma actúan cómo un filtro selectivo cuando se encuentra ante una secuencia Chi genuina.
- Carrasco C, Gilhooly NS, Dillingham MS, Moreno-Herrero F. On the mechanism of recombination hotspot scanning during double-stranded DNA break resection. PNAS June 24, 2013.
¿Cuántas bacterias hay en tus manos?
 Vivimos rodeados de microbios. Microbios
buenos, microbios malos y, sobre todo, microbios ni buenos ni malos.
Vivimos rodeados de microbios. Microbios
buenos, microbios malos y, sobre todo, microbios ni buenos ni malos.
Las bacterias son un tipo de microbios tan pequeños que no los podemos ver a simple vista. Ni siquiera usando una lupa. Para poder verlas necesitamos un microscopio. Por eso, hasta que no se inventó el microscopio nadie sabía que existían.
Ahora sabemos que hay un montón de tipos distintos y conocemos muchas de las que viven cerca de nosotros: en el agua, en la tierra o dentro de nosotros mismos. Sí, incluso dentro de nuestras barrigas. Son minúsculas, pero todas juntas pesan casi tanto como un litro de leche.
Aunque no las podamos ver una a una, los científicos saben criarlas y tener tal cantidad de ellas que podemos ver los grupos que forman. A estos grupos, los científicos los llaman colonias.
Un científico llamado Julius Richard Petri inventó un sistema muy sencillo para criar las bacterias: una placa redonda en la que hay una gelatina que tiene los alimentos que las bacterias necesitan. ¿Cuántos grupos de bacterias (colonias) ves que han crecido en esta placa?.
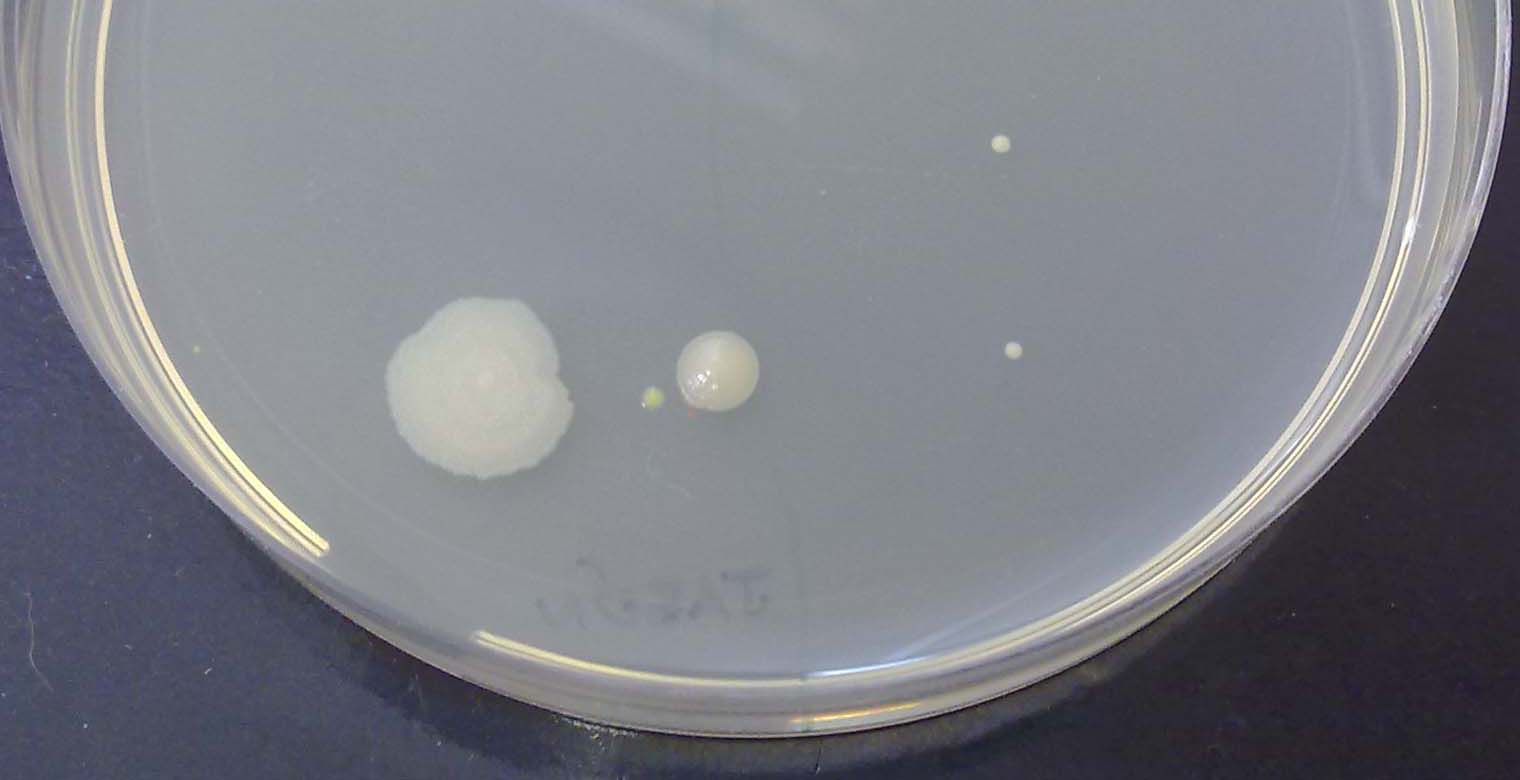
En nuestras manos podemos encontrar un montón de tipos distintos de bacterias. Para verlas, tan solo tenemos que tocar la gelatina de una de las placas de Petri y dejar que crezcan las colonias durante unos días. ¡Tócala con cuidado, no vaya a ser que se rompa!
Si lo haces con las manos limpias, ¿habrá más o menos bacterias? ¿Te apetece hacer el experimento? Toca una mitad de la placa con las manos sucias. Luego lávate las manos y toca la otra mitad. Según pasen los días, observa cómo crecen las colonias.
Como un buen científico, ve anotando durante los días siguientes cuantas colonias salen con las manos sucias y cuantas con ellas limpias.
El último día compara tus resultados con los del resto de la clase y pensar entre todos qué les pasa a las bacterias cuando te lavas las manos.
Una vez que hayan terminado todos los grupos, hay en poner en común los resultados obtenidos y sumar los números de todos y cada una de las parejas.
Esta práctica está basada en una visita que nos hicieron a principios de este año los alumnos del Colegio Rafael Alberti de Coslada. Si te interesa, esta sencilla práctica dirigida a alumnos educación primaria se puede descargar en pdf (2,37 MB).
Programa Internacional de doctorado La Caixa / CNB 2013
Por sexto año consecutivo, la Fundación "la Caixa" convoca su Programa Internacional de doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC.
 Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas para cursar estudios de postgrado tanto en España como en el extranjero.
Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas para cursar estudios de postgrado tanto en España como en el extranjero.
En este sexto año consecutivo, la Fundación "la Caixa" convoca dos contratos predoctorales de personal investigador en formación para desarrollar un doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC, el mayor centro español y uno de los centros líderes en el mundo en el área de Biotecnología, Biología Molecular y Biomedicina.
Las bases de la convocatoria de este año, publicadas en el Boletín Oficial del Estado del 24 de junio de 2013, pueden descargarse como documento pdf (4,61 MB).
Los estudiantes deberán aportar la documentación acreditativa de estar admitido en un programa de doctorado expedido por el responsable de dicho programa.
Las solicitudes pueden enviarse hasta el 1 de julio de 2013.
- Listado provisional de admitidos y excluidos (subsanación de errores hasta el 13 de julio de 2013).
- Listado definitivo de admitidos y excluidos.
- Valoración provisional de méritos del concurso.
- Listado definitivo.
- Resolución definitiva.
España ya forma parte de la infraestructura europea de biología estructural
A través del Centro Nacional de Biotecnología del CSIC, que alberga la unidad de procesamiento de imágenes en microscopía electrónica líder en Europa, España se ha integrado en la mayor infraestructura europea de investigación en biología estructural (Instruct, en sus siglas en inglés).
 Gracias al acuerdo firmado entre el Consejo Superior de Investigaciones Científicas (CSIC) e Instruct, la comunidad científica española tendrá acceso a una red de infraestructuras científicas con capacidades en tecnologías clave para el estudio de la biología estructural de Alemania, Bélgica, Francia, Israel, Italia, Holanda, Portugal, Reino Unido y República Checa. Además, los españoles podrán participar activamente y de forma prioritaria en la preparación y puesta en marcha de nuevos proyectos europeos.
Gracias al acuerdo firmado entre el Consejo Superior de Investigaciones Científicas (CSIC) e Instruct, la comunidad científica española tendrá acceso a una red de infraestructuras científicas con capacidades en tecnologías clave para el estudio de la biología estructural de Alemania, Bélgica, Francia, Israel, Italia, Holanda, Portugal, Reino Unido y República Checa. Además, los españoles podrán participar activamente y de forma prioritaria en la preparación y puesta en marcha de nuevos proyectos europeos.
Los científicos españoles podrán participar activamente y acceder a múltiples infraestructuras Europeas para el desarrollo de sus proyectos de investigación. “El objetivo de esta iniciativa es unir los esfuerzos de varios países que comparten sus instalaciones para avanzar en biología y biomedicina. Los investigadores tendremos acceso a tecnologías de vanguardia, desde sincrotrones hasta grandes instalaciones de resonancia magnética nuclear”, ha señalado el investigador del CSIC José María Carazo.
El Centro Nacional de Biotecnología del CSIC coordina el proyecto en el ámbito del procesamiento de imagen en microscopía electrónica y una de sus misiones será involucrar a diversas instituciones y potenciar sinergias con otros centros de investigación e instalaciones singulares, como por ejemplo, el sincrotrón ALBA, el acelerador de partículas instalado en Cerdanyola del Vallès (Barcelona).
“Supone un gran reconocimiento internacional y nuestro compromiso será el desarrollar la nueva generación de infraestructuras en procesamiento de imagen en microscopía electrónica. Proporcionaremos apoyo a los biólogos experimentales y ayudaremos a maximizar la obtención de conocimiento biológico a partir de imágenes de microscopía. Es todo un honor y un reto el saber que la tecnología que diseñe nuestro grupo será la que se use a nivel europeo en el marco de Instruct”, ha precisado Carazo.
Instruct, la infraestructura europea de investigación en biología estructural, es una de las 48 que forman parte de la hoja de ruta del Foro Estratégico Europeo para las Infraestructuras de Investigación (ESFRI, en sus siglas en inglés). Estas grandes infraestructuras científicas internacionales constituyen la columna vertebral del Espacio Europeo de Investigación y son motor del desarrollo económico de los países involucrados en su construcción, favoreciendo la creación de economías más competitivas e impulsando la recuperación económica en momentos de crisis.
CNB workshop 'Division from A to Z'
7 June 2013
THE WORKSHOP: The divisome is an assembly of proteins involved in bacterial division. Some of its components, as FtsA and FtsZ, share structural and functional homologies with cytoskeletal proteins. Procedures to reconstruct divisomes in the test tube provide further insights on division and supply new discovery platforms to find new antibiotics.
The purpose of the workshop is to discuss the advances in the study of bacterial division.
KEYNOTE SPEAKER: From his postdoctoral at the University of Edinburgh to his present position at the University of Kansas Medical School, Professor Joe Lutkenhaus pioneering work has been crucial for the discovery and study of divisome components. He has received several academic recognitions, among them the prestigious Louisa Gross Horwitz Prize in 2012.
THE PROGRAMME: We have gathered Spanish microbiologists working on bacterial cell division and some investigators whose work, coming from fields different from microbiology, provide important advances to study bacterial division in the light of novel synthetic biology approaches.
VENUE: At the main CNB lecture room. C/ Darwin nº3. 28049 Madrid. Tel: +34 91 585 45 00
Attendance is free but room capacity is limited.
ORGANIZATION: Miguel Vicente (scientific) and Moira Torrent (technical)
WORKSHOP PROGRAMME (pdf)
10:00 - 10:30: José M. Valpuesta (CNB, Madrid) The folding pathway of actin and tubulin mediated by an assembly line of molecular chaperones
10:30 - 11:00: Luis M. Liz-Marzán (CIC biomaGUNE) Nanoplasmonic Biodetection
11:00 - 11:30: Miguel A. de Pedro (CBMSO, Madrid) Cell wall elongation and septation: Who leads whom?
11:30 - 12:00: Juan Ayala (CBMSO, Madrid) Endopeptidases and carboxypeptidases in bacterial cell cycle. How many and for what purpose?
12:00 -12:30 COFFEE BREAK
12:30 - 13:15: Joe Lutkenhaus (U. Kansas) Min system and the Z ring
13:15 - 13:45: Jesús Mingorance (Hospital La Paz, Madrid) FtsA, bacterial division and the quest for a role
14:00 - 15:30 LUNCH
15:30 - 16:00: Marisela Vélez (ICP, Madrid) Looking at individual FtsZ filaments on surfaces in solution: what we have learned so far
16:00 - 16:30: Germán Rivas (CIB, Madrid) Macromolecular interactions of FtsZ: from physical biochemistry to reconstructing minimal divisomes in cell-like environments
16:30 - 17:00: José M. Andreu (CIB, Madrid) Targeting bacterial cell division protein FtsZ with small molecules
17:00 - 17:30: Paulino Gómez-Puertas (CBMSO, Madrid) In silico design of new antimicrobial compounds directed against FtsZ
17:30 - 18:00: Francisco Monroy (U. Complutense, Madrid) Membrane mechanics of bacterial division: a connection to Z ring mechanistic
18:00 - 18:30: Miguel Vicente (CNB, Madrid) Zipping the Z
COOKIES POLICY
A cookie is a text file that is stored on your computer or mobile device via a web server and only that server will be able to retrieve or read the contents of the cookie and allow the Web site remember browser preferences and navigate efficiently. Cookies make the interaction between the user and the website faster and easier.
General information
This Website uses cookies. Cookies are small text files generated by the web pages you visit, which contain the session data that can be useful later in the website. In this way this Web remembers information about your visit, which can facilitate your next visit and make the website more useful.
How do cookies?
Cookies can only store text, usually always anonymous and encrypted. No personal information is ever stored in a cookie, or can be associated with identified or identifiable person.
The data allow this website to keep your information between the pages, and also to discuss how to interact with the website. Cookies are safe because they can only store information that is put there by the browser, which is information the user entered in the browser or included in the page request. You can not run the code and can not be used to access your computer. If a website encrypts cookie data, only the website can read the information.
What types of cookies used?
The cookies used by this website can be distinguished by the following criteria:
1. Types of cookies as the entity that manages:
Depending on who the entity operating the computer or domain where cookies are sent and treat the data obtained, we can distinguish:
- Own cookies: are those that are sent to the user's terminal equipment from a computer or domain managed by the editor itself and from which provides the service requested by the user.
- Third party cookies: these are those that are sent to the user's terminal equipment from a machine or domain that is not managed by the publisher, but by another entity data is obtained through cookies.
In the event that the cookies are installed from a computer or domain managed by the editor itself but the information collected by these is managed by a third party can not be considered as party cookies.
2. Types of cookies as the length of time that remain active:
Depending on the length of time that remain active in the terminal equipment can be distinguished:
- Session cookies: cookies are a type designed to collect and store data while the user accesses a web page. Are usually used to store information that only worth preserving for the service requested by the user at any one time (eg a list of products purchased).
- Persistent cookies: cookies are a type of data which are stored in the terminal and can be accessed and treated for a period defined by the head of the cookie, and can range from a few minutes to several years.
3. Cookies types according to their purpose:
Depending on the purpose for which the data are processed through cookies, we can distinguish between:
- Technical cookies: these are those that allow the user to navigate through a web page or application platform and the use of different options or services it exist as, for example, control traffic and data communication, identify the session, access to restricted access parts, remember the elements of an order, make the buying process an order, make an application for registration or participation in an event, use security features while browsing store content for dissemination videos or sound or share content via social networks.
- Customization cookies: these are those that allow the user to access the service with some general characteristics based on a predefined set of criteria in the user terminal would eg language, the type of browser through which you access the service, the locale from which you access the service, etc.
- Analysis cookies: they are those that allow the responsible for them, monitoring and analyzing the behavior of users of the web sites that are linked. The information gathered through such cookies are used in measuring the activity of web sites, application or platform and for the profiling of user navigation of such sites, applications and platforms, in order to make improvements function data analysis how users use the service.
Management tool cookies
This Website uses Google Analytics.
Google Analytics is a free tool from Google that primarily allows website owners know how users interact with your website. Also, enable cookies in the domain of the site in which you are and uses a set of cookies called "__utma" and "__utmz" to collect information anonymously and reporting of website trends without identifying individual users..
For statistics of use of this website use cookies in order to know the level of recurrence of our visitors and more interesting content. This way we can concentrate our efforts on improving the most visited areas and make the user more easily find what they are looking for. On this site you can use the information from your visit for statistical evaluations and calculations anonymous data and to ensure the continuity of service or to make improvements to their websites. For more details, see the link below privacy policy [http://www.google.com/intl/en/policies/privacy/]
How to manage cookies on your computer: disabling and deleting cookies
All Internet browsers allow you to limit the behavior of a cookie or disable cookies within settings or browser settings. The steps for doing so are different for each browser, you can find instructions in the help menu of your browser.
If you decline the use of cookies, since it is possible thanks to the preferences menu of your browser or settings, reject, this website will continue to function properly without the use of the same.
Can you allow, block or delete cookies installed on your computer by setting your browser options installed on your computer:
- For more information about Internet Explorer click here.
- For more information on Chrome click here.
- For more information about Safari click here.
- For more information about Firefox click here.
Through your browser, you can also view the cookies that are on your computer, and delete them as you see fit. Cookies are text files, you can open and read the contents. The data within them is almost always encrypted with a numeric key corresponding to an Internet session so often has no meaning beyond the website who wrote it.
Informed consent
The use of this website on the other hand, implies that you paid your specific consent to the use of cookies, on the terms and conditions provided in this Cookies Policy, without prejudice to the measures of deactivation and removal of cookies that you can take, and mentioned in the previous section.





