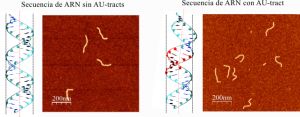
Identificadas las secuencias que aportan flexibilidad a la molécula de ARN
Roberto Solano consolida su posición como uno de los científicos más citados

Roberto Solano, investigador y actual director del Departamento de Genética Molecular de Plantas en el Centro Nacional de Biotecnología vuelve a aparecer en la lista de los investigadores más citados en el área de ciencias animales y vegetales publicada por Clarivate Analytics.
Solano se encuentra, por séptimo año consecutivo, entre los más de 100 científicos españoles reconocidos este año, y entre los 17 pertenecientes al Consejo Superior de Investigaciones Científicas.
Esta clasificación reconoce a más de 6000 investigadores que han contribuido de manera excepcional al conocimiento científico en su área de trabajo gracias a la publicación de numerosos artículos con un alto índice de impacto entre los años 2009 y 2019 y que, además, se encuentran dentro del 1% de los más citados a nivel global.
El laboratorio del Dr. Solano estudia los mecanismos moleculares que permiten a las plantas adaptarse al medio ambiente, centrándose en el funcionamiento del jasmonato, una hormona imprescindible para responder al estrés. Sus trabajos han llevado al descubrimiento de la forma activa de esta hormona y la de una familia de proteínas imprescindibles para que el jasmonato ejerza su acción, tanto en Arabidopsis thaliana como en Marchantia polymorpha. Estos trabajos se han visto reflejados en el año 2020 en publicaciones en ‘Current Biology’, ‘Plant Journal’ y ‘PNAS’.

XXVIII Workshop AVANCES EN BIOLOGÍA MOLECULAR POR JÓVENES INVESTIGADORES EN EL EXTRANJERO
Noche de los Investigadores 2020

Con motivo de la celebración de la Noche de los Investigadores e Investigadoras el viernes 27 de noviembre de 2020, los centros del Consejo Superior de Investigaciones Científicas (CSIC) del campus de Cantoblanco organizamos un evento online con Medialab Prado. Hay actividades abiertas al público general y familiar en horario de tarde (17:00-21:00) y otras dirigidas a centros educativos en horario de mañana (9:30-14:30).
Encontraréis talleres y demostraciones científicas que acercan a la investigación en ciencia de materiales, biotecnología, tecnología de los alimentos, tecnologías químicas y matemáticas que se realiza en estos centros, junto con talleres de ciencia ciudadana de Medialab Prado. También habrá una gymkana para conocer los descubrimientos de grandes científicas pioneras. Todas las actividades requieren reserva previa, que se habilitará a partir del 16 de noviembre.
- Actividades dirigidas a Centros Educativos. Horario (9:30-14:30)
- Actividades Público General. Horario Tarde (17:00-21:00)
Centros CSIC participantes: Instituto de Ciencia de Materiales de Madrid (ICMM), Instituto de Catálisis y Petroleoquímica (ICP), Centro Nacional de Biotecnología (CNB), Instituto de Investigación en Ciencias de la Alimentación (CIAL), Instituto de Cerámica y Vidrio (ICV), Instituto de Ciencias Matemáticas (ICMAT), Instituto de Física Teórica (IFT), Instituto de Micro y Nanotecnología (IMN), Centro de Biología Molecular Severo Ochoa (CBM).
Medialab Prado: laboratorio ciudadano https://www.medialab-prado.es/


El CNB y el CSIC, XXII Premios Derechos Humanos de la Abogacía en la categoría de Instituciones

- El director de la OMS Tedros Adhanom, Jesús Blasco de Avellaneda y Elena Arce también son galardoandos en la XXII edición de los Premios Derechos Humanos de la Abogacía
El Consejo General de la Abogacía Española a través de la Fundación Abogacía ha otorgado al Consejo Superior de Investigaciones Científicas (CSIC) y su Centro Nacional de Biotecnología (CNB-CSIC) el XXII Premio Derechos Humanos en la categoría de “Instituciones”, por ser el mayor referente en nuestro país en la investigación científica y la búsqueda de avances que ayuden a prevenir y curar enfermedades. La pandemia de Covid-19 no ha hecho más que confirmar el excelente trabajo de ambas instituciones, que han desarrollado un gran número de proyectos de investigación orientados a desarrollar vacunas, compuestos antivirales y terapéuticos, modelos y medidas para reducir el contagio e identificación de personas positivas.
Además del reconocimiento a la labor del CNB y el CSIC, el Consejo General de la Abogacía Española ha premiado a Tedros Adhanom Ghebreyesus, director de la Organización Mundial de la Salud como cara visible de una organización que ha estado en primera línea mundial desde que se declaró la pandemia mundial provocada por el Covid-19; Jesús Blasco de Avellaneda, fotoperiodista, por la visibilización de las vulneraciones de los derechos fundamentales de los colectivos más desfavorecidos y Elena Arce, por su defensa incansable de los derechos de los menores extranjeros no acompañados.
Más información:
Un estudio examina la interacción de las proteínas del coronavirus y la célula infectada para lograr dianas terapéuticas
Una nueva metodología permite la caracterización de consorcios bacterianos
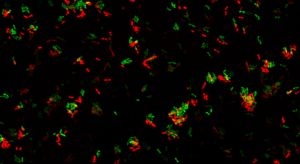
- La formación de consorcios permite a las comunidades bacterianas mejorar su supervivencia, y esta característica tiene un alto interés en el desarrollo de consorcios artificiales con aplicaciones industriales
- Un equipo de investigadores del CNB-CSIC describe una nueva metodología para caracterizar la composición de este tipo de agregados bacterianos.
En la naturaleza, algunos microorganismos (bacterias y hongos) forman estructuras biológicas con un alto grado de organización estructural y capacidad de albergar comunidades formadas por múltiples especies distintas. Estas son denominadas formalmente biopelículas (biofilms). Dichas comunidades microbianas poseen una alta capacidad de sobrevivir en el medioambiente en el que habitan: se protegen mediante la secreción de complejos cócteles químicos que las permiten adherirse fuertemente a superficies y tejidos, así como resistir mucho más cualquier agresión externa. Además, pueden producirse acciones simbióticas/cooperativas entre las diferentes especies de microorganismos que les permitan mejorar su supervivencia compartiendo nutrientes y especializándose cada una de las especies en la realización de funciones esenciales para el consorcio.
Esta característica observada en los consorcios microbianos puede ser de gran interés en biotecnología, donde la utilización de técnicas de ingeniería y biología sintética podría habilitar la explotación de sus características para el desarrollo de aplicaciones industriales (como por ejemplo la producción de sustratos enzimáticos o la descontaminación de aguas residuales).
Uno de los retos del desarrollo de estos nuevos consorcios artificiales es la caracterización de su estructura y de la heterogeneidad de su composición mediante parámetros cuantificables. Investigadores del Consejo Superior de Investigaciones Científicas liderados por Víctor de Lorenzo en el Centro Nacional de Biotecnología (CNB-CSIC) publican ahora en la revista Water Research una nueva metodología basada en el uso de parámetros que analizan la composición y morfología de los agregados de manera precisa.
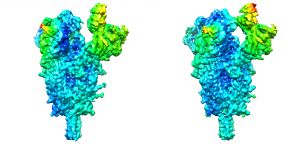
Nuevos análisis computaciones determinan la flexibilidad de la proteína de la espícula (Spike) del SARS-CoV2
El investigador del CNB Esteban Veiga recibe una Beca FERO para la investigación en Cáncer de Mama

- La Fundación FERO entrega una de las Becas de este año al investigador del CNB Esteban Veiga.
- Su proyecto se centra en la investigación de nuevas inmunoterapias contra el cáncer de mama.
La Fundación FERO ha anunciado los ganadores de las XIX Becas FERO y el ganador del II Proyecto FERO-Mango de investigación en Cáncer de Mama, en un encuentro virtual que tuvo lugar el pasado 19 de octubre de 2020. Las becas, de 80.000€ cada una, están destinadas a ayudar a jóvenes investigadores a desarrollar durante dos años el proyecto de investigación traslacional en cáncer presentado.
Esteban Veiga Chacón, investigador del Centro Nacional de Biotecnología, perteneciente al Consejo Superior de Investigaciones Científicas (CNB-CSIC), ha obtenido la Beca Fero gracias a su investigación “Next generation of cancer immunotherapies based in bacteria-trained lymphocytes”. Este proyecto se centra en consolidar los datos obtenidos sobre la eficacia de las células bacT utilizadas como inmunoterapia novedosa para combatir tumores, centrándose concretamente en el cáncer de mama.
“Para poder consolidar las pruebas de concepto realizadas donde demostramos la utilidad de los linfocitos entrenados con bacterias (bacT) como nueva terapia antitumoral utilizaremos diversos tumores mamarios de ratones. Si conseguimos además reactivar las células CAR-T, sería un gran paso hacia nuevas terapias para tumores sólidos, un campo donde esta técnica no ha dado buenos resultados por ahora. Además, el proyecto pretende generar nuevos algoritmos para la identificación de los neoantígenos tumorales, uno de los mayores retos de la inmunoterapia tumoral”, explica el Dr. Esteban Veiga Chacón.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.