Según se van sintetizando las proteínas en los ribosomas, éstas tienen que ir tomando la forma adecuada para poder ejercer su función. Aunque las características físico-químicas de los aminoácidos que las forman determinan en gran parte la forma que adquieren, hay muchas proteínas que necesitan ayuda extra de parte de un grupo de proteínas conocidas como chaperonas.
 Para comprender mejor cómo funcionan estas proteínas, en su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo dirigido por José María Valpuesta ha utilizado la microscopía electrónica. Gracias a esta técnica han podido determinar por primera vez la estructura de un complejo formado por la chaperona DnaJ y su sustrato, lo que les ha permitido observar cómo la chaperona cambia la estructura del sustrato y con ello su función.
Para comprender mejor cómo funcionan estas proteínas, en su laboratorio del Centro Nacional de Biotecnología del CSIC (CNB), el grupo dirigido por José María Valpuesta ha utilizado la microscopía electrónica. Gracias a esta técnica han podido determinar por primera vez la estructura de un complejo formado por la chaperona DnaJ y su sustrato, lo que les ha permitido observar cómo la chaperona cambia la estructura del sustrato y con ello su función.
En colaboración con la Universidad del País Vasco, el investigador postdoctoral del CNB Jorge Cuéllar ha identificado además en dicha chaperona una zona de gran flexibilidad que le permite adaptarse a la forma de distintas proteínas. Como se puede apreciar en la imagen de abajo, la forma que adopta la chaperona DnaJ (en azul) cambia radicalmente en función del sustrato al que se une (RepE1-144, RepE o Rep54; en amarillo). De este modo, una misma chaperona es capaz de unirse a una variedad de proteínas diferentes, consiguiendo en todas ellas que adquieran la forma necesaria para funcionar.
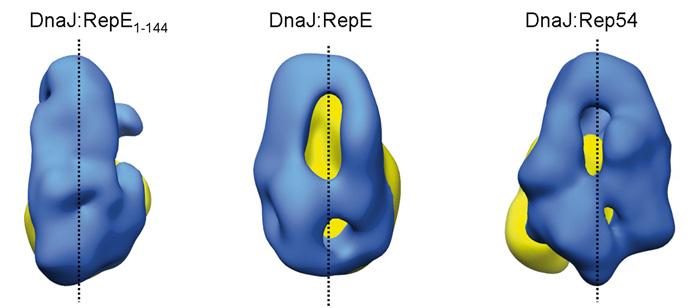
- Cuéllar J, Perales-Calvo J, Muga A, Valpuesta JM, Moro F. Structural insights into the chaperone activity of the 40 kDa heat shock protein DnaJ. Binding and remodeling of a native substrate. J Biol Chem 2013 Apr 11. [doi: 10.1074/jbc.M112.430595].
Nucleic Acids Res. 2013 Feb 1;41(3):1711-21
Seco EM, Zinder JC, Manhart CM, Lo Piano A, McHenry CS, Ayora S.
 Complex viruses that encode their own initiation proteins and subvert the host's elongation apparatus have provided valuable insights into DNA replication. Using purified bacteriophage SPP1 and Bacillus subtilis proteins, we have reconstituted a rolling circle replication system that recapitulates genetically defined protein requirements.
Complex viruses that encode their own initiation proteins and subvert the host's elongation apparatus have provided valuable insights into DNA replication. Using purified bacteriophage SPP1 and Bacillus subtilis proteins, we have reconstituted a rolling circle replication system that recapitulates genetically defined protein requirements.
Eleven proteins are required: phage-encoded helicase (G40P), helicase loader (G39P), origin binding protein (G38P) and G36P single-stranded DNA-binding protein (SSB); and host-encoded PolC and DnaE polymerases, processivity factor (β(2)), clamp loader (τ-δ-δ') and primase (DnaG). This study revealed a new role for the SPP1 origin binding protein. In the presence of SSB, it is required for initiation on replication forks that lack origin sequences, mimicking the activity of the PriA replication restart protein in bacteria. The SPP1 replisome is supported by both host and viral SSBs, but phage SSB is unable to support B. subtilis replication, likely owing to its inability to stimulate the PolC holoenzyme in the B. subtilis context. Moreover, phage SSB inhibits host replication, defining a new mechanism by which bacterial replication could be regulated by a viral factor.
Sánchez-Sampedro L, Gómez CE, Mejías-Pérez E, Pérez-Jiménez E, Oliveros JC, Esteban M.
 Replication competent poxvirus vectors with an attenuation phenotype and with high immunogenic capacity of the foreign expressed antigen are being pursuit as novel vaccine vectors against different pathogens. In this investigation we have examined the replication and immunogenic characteristics of two vaccinia virus (VACV) mutants M65 and M101.
Replication competent poxvirus vectors with an attenuation phenotype and with high immunogenic capacity of the foreign expressed antigen are being pursuit as novel vaccine vectors against different pathogens. In this investigation we have examined the replication and immunogenic characteristics of two vaccinia virus (VACV) mutants M65 and M101.
These mutants were generated after 65 and 101 serial passages of persistently infected Friend erythroleukemia cells (FEL). In cultured cells of different origins the mutants are replication competent and have growth kinetics similar or slightly reduced in comparison with the parental Western Reserve (WR) virus strain. In normal and immune suppressed infected mice the mutants showed different attenuation levels, and pathogenicity in comparison with WR and MVA strains. Wide genome analysis after deep sequencing revealed selected genomic deletions and mutations in a number of viral open-reading frames (ORFs). Mice immunized in DNA prime/Mutant boost regime with viral vectors expressing the LACK antigen of Leismania infantum resulted in protection or delay in the onset of cutaneus leishmaniasis. Protection was similar to that triggered by MVA-LACK. In immunized mice, both polyfunctional CD4+ and CD8+ T cells with an effector memory phenotype were activated by the two mutants, but DNA-LACK/M65-LACK protocol preferentially induced CD4+ while DNA-LACK/M101-LACK preferentially induced CD8+ T cell responses.
Altogether our findings showed the adaptive changes of the WR genome during long-term virus-host cell interaction and how replication competency of M65 and M101 mutants confers distinct biological properties and immunogenicity in mice, as compared to the MVA strain. These mutants could have applicability for understanding VACV biology and as potential vaccine vectors against pathogens and tumors.
J Virol. 2013 Jul;87(13):7282-300
Gómez CE, Perdiguero B, Cepeda MV, Mingorance L, García-Arriaza J, Vandermeeren A, Sorzano CO, Esteban M.
 A major goal in the control of hepatitis C infection is the development of a vaccine. Here, we have developed a novel HCV vaccine candidate based on the highly attenuated poxvirus vector MVA (referred as MVA-HCV) expressing the near full-length (7.9 kbp) HCV sequence, with the aim to target almost all of the T and B cell determinants described for HCV.
A major goal in the control of hepatitis C infection is the development of a vaccine. Here, we have developed a novel HCV vaccine candidate based on the highly attenuated poxvirus vector MVA (referred as MVA-HCV) expressing the near full-length (7.9 kbp) HCV sequence, with the aim to target almost all of the T and B cell determinants described for HCV.
In infected cells, MVA-HCV produces a polyprotein that is subsequently processed into the structural and nonstructural HCV proteins, triggering the cytoplasmic accumulation of dense membrane aggregates. In both C57BL/6 and transgenic HLA-A2 vaccinated mice, MVA-HCV induced high, broad, polyfunctional and long-lasting HCV-specific T cell immune responses. The vaccine-induced T cell response was mainly mediated by CD8 T cells; however, although lower in magnitude, the CD4+ T cells were highly polyfunctional. In homologous protocol (MVA-HCV/MVA-HCV) the main CD8+ T cell target was p7+NS2, whereas in heterologous combination (DNA-HCV/MVA-HCV) the main target was NS3. Antigenic responses were also detected against other HCV proteins (Core, E1-E2, NS4) but the magnitude of the responses was dependent of the protocol used. The majority of the HCV-induced CD8+ T cells were triple or quadruple-cytokine producers. The MVA-HCV vaccine induced memory CD8+ T cell responses with an effector memory (TEM) phenotype.
Overall, our data showed that MVA-HCV induced broad, highly polyfunctional and durable T cell responses of a magnitude and quality that might be associated with protective immunity and open the path for future considerations of MVA-HCV as prophylactic and/or therapeutic vaccine candidate against HCV.
Un estudio internacional en el que ha participado la investigadora del CNB Cristina Risco ha publicado en la revista PNAS la estructura de la ribonucleoproteína del virus Bunyamwera, modelo de estudio de los integrantes de la familia Bunyaviridae, a la que también pertenecen los virus que provocan la fiebre hemorrágica de Congo y Crimea y la fiebre del valle del Rift.
"Uno de los principales resultados de esta investigación es que la flexibilidad de la nucleoproteína que envuelve el ARN de Bunyamwera facilita el empaquetamiento de su material genético en las partículas virales y su replicación una vez fuera del virus. La estructura atómica muestra que el plegamiento de la nucleoproteína es, además, diferente al observado en las demás nucleoproteínas de virus conocidas”, explica Risco.
Este estudio muestra que, a pesar de su flexibilidad, el complejo que recubre el material genético lo protege completamente del ataque de las nucleasas, las enzimas que rompen las moléculas de ARN.
Los resultados de este estudio ayudan a entender mejor el mecanismo molecular mediante el que se ensamblan las ribonucleoproteínas virales. "Este conocimiento permitirá avanzar en el desarrollo de compuestos para atacar a estos complejos macromoleculares que son esenciales para la supervivencia de los virus ARN", añade desde el mismo laboratorio del Centro Nacional de Biotecnología del CSIC la investigadora Isabel Fernández de Castro.
Los hallazgos logrados por este estudio, liderado por investigadores chinos, ha sido posible mediante el uso de cristalografía de rayos X y microscopía electrónica. Como se aprecia en la imagen, el complejo formado por el monómero de la proteína viral NP y el ARN, la flexibilidad de este complejo respecto a los adyacentes es el elemento básico que dirige la oligomerización y ensamblaje de las ribonucleoproteínas lineales y flexibles del virus Bunyamwera.
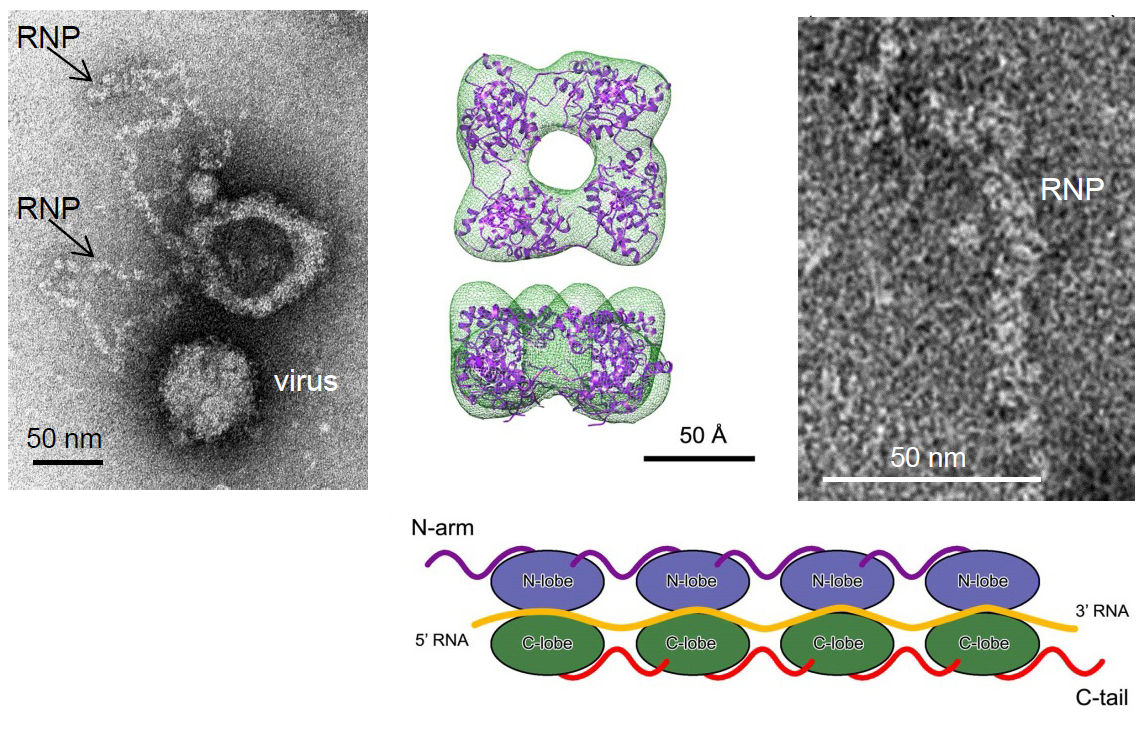
- Li B, Wang Q, Pan X, Fernández de Castro I, Sun Y, Guo Y, Tao X, Risco C, Su SF, Lou Z. Bunyamwera virus possesses a distinct nucleocapsid protein to facilitate genome encapsidation. PNAS April 8, 2013 doi: 10.1073/pnas.1222552110.
Li B, Wang Q, Pan X, Fernández de Castro I, Sun Y, Guo Y, Tao X, Risco C, Su SF, Lou Z.
 Bunyamwera virus (BUNV), which belongs to the genus Orthobunyavirus, is the prototypical virus of the Bunyaviridae family. Similar to other negative-sense single-stranded RNA viruses, bunyaviruses possess a nucleocapsid protein (NP) to facilitate genomic RNA encapsidation and virus replication.
Bunyamwera virus (BUNV), which belongs to the genus Orthobunyavirus, is the prototypical virus of the Bunyaviridae family. Similar to other negative-sense single-stranded RNA viruses, bunyaviruses possess a nucleocapsid protein (NP) to facilitate genomic RNA encapsidation and virus replication.
The structures of two NPs of members of different genera within the Bunyaviridae family have been reported. However, their structures, RNA-binding features, and functions beyond RNA binding significantly differ from one another. Here, we report the crystal structure of the BUNV NP–RNA complex. The polypeptide of the BUNV NP was found to possess a distinct fold among viral NPs. An N-terminal arm and a C-terminal tail were found to interact with neighboring NP protomers to form a tetrameric ring-shaped organization. Each protomer bound a 10-nt RNA molecule, which was acquired from the expression host, in the positively charged crevice between the N and C lobes. Inhomogeneous oligomerization was observed for the recombinant BUNV NP–RNA complex, which was similar to the Rift Valley fever virus NP–RNA complex.
This result suggested that the flexibility of one NP protomer with adjacent protomers underlies the BUNV ribonucleoprotein complex (RNP) formation. Electron microscopy revealed that the monomer-sized NP–RNA complex was the building block of the natural BUNV RNP. Combined with previous results indicating that mutagenesis of the interprotomer or protein–RNA interface affects BUNV replication, our structure provides a great potential for understanding the mechanism underlying negative-sense single-stranded RNA RNP formation and enables the development of antiviral therapies targeting BUNV RNP formation.
Castón JR, Luque D, Gómez-Blanco J, Ghabrial SA.
 Chrysoviruses are double-stranded RNA viruses with a multipartite genome. Structure of two fungal chrysoviruses, Penicillium chrysogenum virus and Cryphonectria nitschkei chrysovirus 1, has been determined by three-dimensional cryo-electron microscopy analysis and in hydrodynamic studies.
Chrysoviruses are double-stranded RNA viruses with a multipartite genome. Structure of two fungal chrysoviruses, Penicillium chrysogenum virus and Cryphonectria nitschkei chrysovirus 1, has been determined by three-dimensional cryo-electron microscopy analysis and in hydrodynamic studies.
The capsids of both viruses are based on a T=1 lattice containing 60 subunits, remain structurally undisturbed throughout the viral cycle, and participate in genome metabolism. The capsid protein is formed by a repeated α-helical core, indicative of gene duplication.
Whereas the chrysovirus capsid protein has two motifs with the same fold, most dsRNA virus capsid subunits consist of dimers of a single protein with similar folds. The arrangement of the chrysovirus α-helical core is conserved in the totivirus L-A capsid protein, suggesting a shared basic fold. The encapsidated genome is organized in concentric shells; whereas inner dsRNA shells are diffuse, the outermost layer is organized into a dodecahedral cage beneath the protein capsid. This genome ordering could constitute a framework for dsRNA transcription in the capsid interior and/or have a structural role for capsid stability.
El Campus de Excelencia Internacional UAM+CSIC (CEI UAM+CSIC) tiene como una de sus líneas de actuación esenciales la captación de talento tanto a nivel nacional como internacional. Y con la intención de atraer científicos y profesores de primer nivel, acaba de convocar el Premio de Investigación CEI UAM+CSIC en el área de la Biología Molecular Física y Sintética.
 El objetivo de este premio, compatible con cualquier otro tipo de premio y/o ayuda, es el de favorecer que profesionales de otros centros de investigación puedan trasladarse al CNB para desarrollar una línea de investigación que se centre en el estudio del plegamiento y función de proteínas. Para ello, deberán integrar métodos experimentales de alta resolución temporal, estructural y de moléculas únicas, modelización teórica y simulaciones computacionales.
El objetivo de este premio, compatible con cualquier otro tipo de premio y/o ayuda, es el de favorecer que profesionales de otros centros de investigación puedan trasladarse al CNB para desarrollar una línea de investigación que se centre en el estudio del plegamiento y función de proteínas. Para ello, deberán integrar métodos experimentales de alta resolución temporal, estructural y de moléculas únicas, modelización teórica y simulaciones computacionales.
El premio tiene una dotación económica de 30.000 € anuales de libre disposición durante tres años, prorrogables a otros dos. Para solicitarlo, el investigador debe tener una vinculación permanente con el CSIC y no podrá estar adscrito a ningún centro del CEI UAM+CSIC.
Las solicitudes se presentarán en el registro general de la UAM desde el 8 de abril al 22 de abril de 2013.
Philos Trans R Soc Lond B Biol Sci. 2013 Mar 11;368(1616):20120377
Nikel PI, Pérez-Pantoja D, de Lorenzo V.
 Chlorinated pollutants are hardly biodegradable under oxic conditions, but they can often be metabolized by anaerobic bacteria through organohalide respiration reactions.
Chlorinated pollutants are hardly biodegradable under oxic conditions, but they can often be metabolized by anaerobic bacteria through organohalide respiration reactions.
In an attempt to identify bottlenecks limiting aerobic catabolism of 1,3-dichloroprop-1-ene (1,3-DCP; a widely used organohalide) in Pseudomonas pavonaceae, the possible physiological restrictions for this process were surveyed. Flow cytometry and a bioluminescence reporter of metabolic state revealed that cells treated with 1,3-DCP experienced an intense stress that could be traced to the endogenous production of reactive oxygen species (ROS) during the metabolism of the compound. Cells exposed to 1,3-DCP also manifested increased levels of d-glucose-6-P 1-dehydrogenase activity (G6PDH, an enzyme key to the synthesis of reduced NADPH), observed under both glycolytic and gluconeogenic growth regimes. The increase in G6PDH activity, as well as cellular hydroperoxide levels, correlated with the generation of ROS. Additionally, the high G6PDH activity was paralleled by the accumulation of d-glucose-6-P, suggesting a metabolic flux shift that favours the production of NADPH. Thus, G6PDH and its cognate substrate seem to play an important role in P. pavonaceae under redox stress caused by 1,3-DCP, probably by increasing the rate of NADPH turnover.
The data suggest that oxidative stress associated with the biodegradation of 1,3-DCP reflects a significant barrier for the evolution of aerobic pathways for chlorinated compounds, thereby allowing for the emergence of anaerobic counterparts.
In this paper we have addressed the problem of analysing Next Generation Sequencing samples with an expected large biodiversity content. We analysed several well-known 16S rRNA datasets from experimental samples, including both large and short sequences, in numbers of tens of thousands, in addition to carefully crafted synthetic datasets containing more than 7000 OTUs.
From this data analysis several patterns were identified and used to develop new guidelines for experimentation in conditions of high biodiversity. We analysed the suitability of different clustering packages for these type of situations, the problem of even sampling, the relative effectiveness of Chao1 and ACE estimators as well as their effect on sampling size for a variety of population distributions. As regards practical analysis procedures, we advocated an approach that retains as much high-quality experimental data as possible. By carefully applying selection rules combining the taxonomic assignment with clustering strategies, we derived a set of recommendations for ultra-sequencing data analysis at high biodiversity levels.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.





