Carrasco C, Gilhooly NS, Dillingham MS, Moreno-Herrero F.
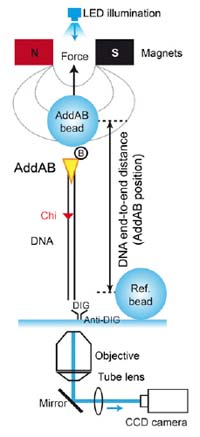 We report the use of the magnetic tweezers approach to monitor the dynamics of double-stranded DNA break resection at single-molecule resolution. In this work, we unveil the inner workings of the Bacillus subtilis AddAB complex by showing that the domain responsible for the recognition of the recombination hotspot sequence Crossover hotspot instigator (Chi) antagonizes the translocation activity, causing the complex to transiently stall at Chi and Chi-like sequences.
We report the use of the magnetic tweezers approach to monitor the dynamics of double-stranded DNA break resection at single-molecule resolution. In this work, we unveil the inner workings of the Bacillus subtilis AddAB complex by showing that the domain responsible for the recognition of the recombination hotspot sequence Crossover hotspot instigator (Chi) antagonizes the translocation activity, causing the complex to transiently stall at Chi and Chi-like sequences.
We propose a simple model for failed and successful Chi recognition in which the battle between the translocation and sequence-specific binding activities acts as a selectivity filter for bona fide Chi sequences.
 Vivimos rodeados de microbios. Microbios
buenos, microbios malos y, sobre todo, microbios ni buenos ni malos.
Vivimos rodeados de microbios. Microbios
buenos, microbios malos y, sobre todo, microbios ni buenos ni malos.
Las bacterias son un tipo de microbios tan pequeños que no los podemos ver a simple vista. Ni siquiera usando una lupa. Para poder verlas necesitamos un microscopio. Por eso, hasta que no se inventó el microscopio nadie sabía que existían.
Ahora sabemos que hay un montón de tipos distintos y conocemos muchas de las que viven cerca de nosotros: en el agua, en la tierra o dentro de nosotros mismos. Sí, incluso dentro de nuestras barrigas. Son minúsculas, pero todas juntas pesan casi tanto como un litro de leche.
Aunque no las podamos ver una a una, los científicos saben criarlas y tener tal cantidad de ellas que podemos ver los grupos que forman. A estos grupos, los científicos los llaman colonias.
Un científico llamado Julius Richard Petri inventó un sistema muy sencillo para criar las bacterias: una placa redonda en la que hay una gelatina que tiene los alimentos que las bacterias necesitan. ¿Cuántos grupos de bacterias (colonias) ves que han crecido en esta placa?.
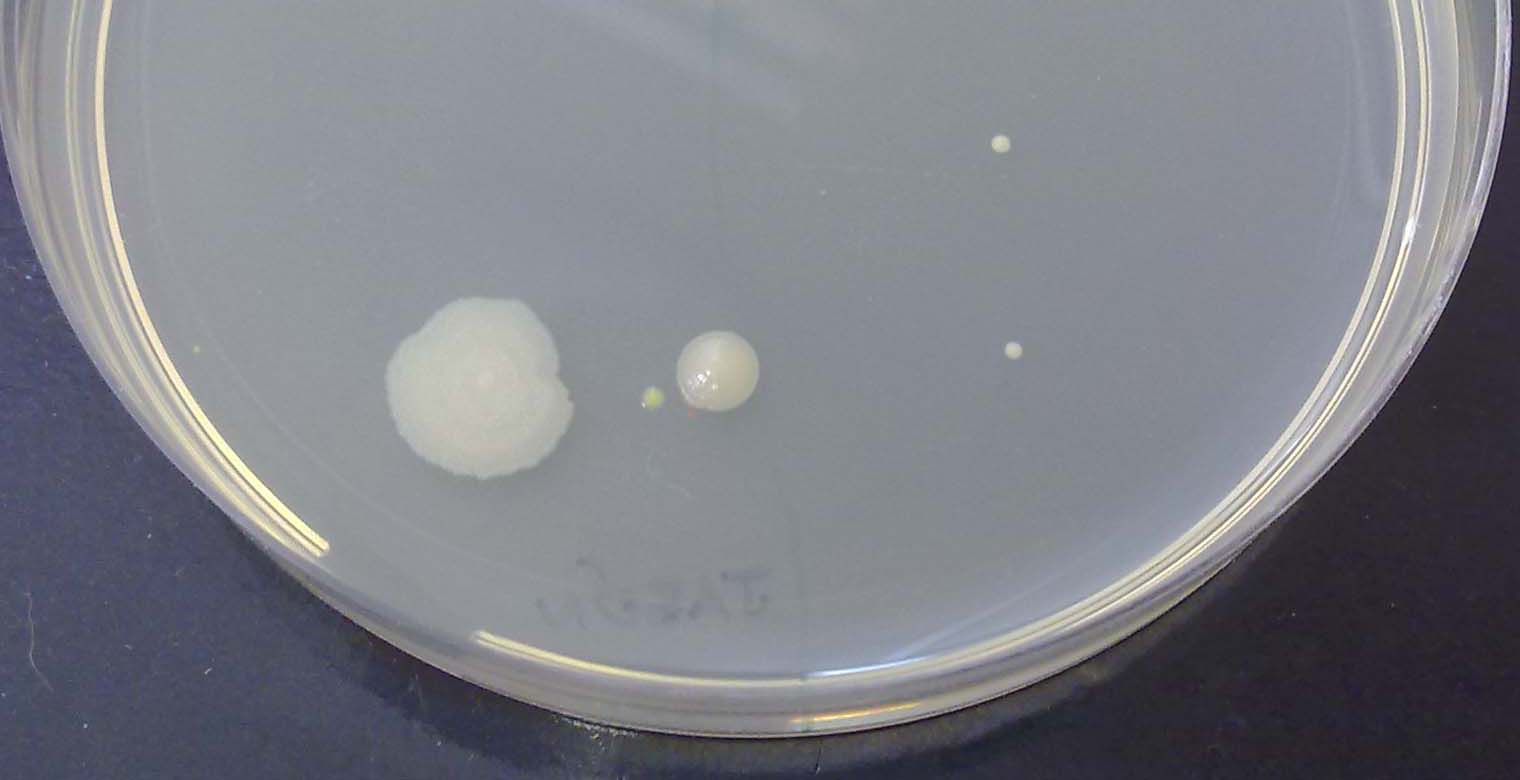
En nuestras manos podemos encontrar un montón de tipos distintos de bacterias. Para verlas, tan solo tenemos que tocar la gelatina de una de las placas de Petri y dejar que crezcan las colonias durante unos días. ¡Tócala con cuidado, no vaya a ser que se rompa!
Si lo haces con las manos limpias, ¿habrá más o menos bacterias? ¿Te apetece hacer el experimento? Toca una mitad de la placa con las manos sucias. Luego lávate las manos y toca la otra mitad. Según pasen los días, observa cómo crecen las colonias.
Como un buen científico, ve anotando durante los días siguientes cuantas colonias salen con las manos sucias y cuantas con ellas limpias.
El último día compara tus resultados con los del resto de la clase y pensar entre todos qué les pasa a las bacterias cuando te lavas las manos.
Una vez que hayan terminado todos los grupos, hay en poner en común los resultados obtenidos y sumar los números de todos y cada una de las parejas.
Esta práctica está basada en una visita que nos hicieron a principios de este año los alumnos del Colegio Rafael Alberti de Coslada. Si te interesa, esta sencilla práctica dirigida a alumnos educación primaria se puede descargar en pdf (2,37 MB).
Mol Cell. 2013 Jun 4. pii: S1097-2765(13)00365-1
Trinidad AG, Muller PA, Cuellar J, Klejnot M, Nobis M, Valpuesta JM, Vousden KH.

We show that folding of wild-type p53 is promoted by an interaction with the chaperonin CCT. Depletion of this chaperone in cells results in the accumulation of misfolded p53, leading to a reduction in p53-dependent gene expression. Intriguingly, p53 proteins mutated to prevent the interaction with CCT show conformational instability and acquire an ability to promote invasion and random motility that is similar to the activity of tumor-derived p53 mutants. Our data therefore suggest that both growth suppression and cell invasion may be differentially regulated functions of wild-type p53.
Ortega-Esteban A, Pérez-Berná AJ, Menéndez-Conejero R, Flint SJ, San Martín C, de Pablo PJ.
 The standard pathway for virus infection of eukaryotic cells requires disassembly of the viral shell to facilitate release of the viral genome into the host cell. Here we use mechanical fatigue, well below rupture strength, to induce stepwise disruption of individual human adenovirus particles under physiological conditions, and simultaneously monitor disassembly in real time.
The standard pathway for virus infection of eukaryotic cells requires disassembly of the viral shell to facilitate release of the viral genome into the host cell. Here we use mechanical fatigue, well below rupture strength, to induce stepwise disruption of individual human adenovirus particles under physiological conditions, and simultaneously monitor disassembly in real time.
Our data show the sequence of dismantling events in individual mature (infectious) and immature (noninfectious) virions, starting with consecutive release of vertex structures followed by capsid cracking and core exposure. Further, our experiments demonstrate that vertex resilience depends inextricably on maturation, and establish the relevance of penton vacancies as seeding loci for virus shell disruption. The mechanical fatigue disruption route recapitulates the adenovirus disassembly pathway in vivo, as well as the stability differences between mature and immature virions.
Por sexto año consecutivo, la Fundación "la Caixa" convoca su Programa Internacional de doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC.
 Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas para cursar estudios de postgrado tanto en España como en el extranjero.
Por su presupuesto y volumen de actividad, la Fundación "la Caixa" figura entre las diez fundaciones más importantes del mundo. Desde 1982, mantiene diversos programas para cursar estudios de postgrado tanto en España como en el extranjero.
En este sexto año consecutivo, la Fundación "la Caixa" convoca dos contratos predoctorales de personal investigador en formación para desarrollar un doctorado en Biomedicina en el Centro Nacional de Biotecnología del CSIC, el mayor centro español y uno de los centros líderes en el mundo en el área de Biotecnología, Biología Molecular y Biomedicina.
Las bases de la convocatoria de este año, publicadas en el Boletín Oficial del Estado del 24 de junio de 2013, pueden descargarse como documento pdf (4,61 MB).
Los estudiantes deberán aportar la documentación acreditativa de estar admitido en un programa de doctorado expedido por el responsable de dicho programa.
Las solicitudes pueden enviarse hasta el 1 de julio de 2013.
- Listado provisional de admitidos y excluidos (subsanación de errores hasta el 13 de julio de 2013).
- Listado definitivo de admitidos y excluidos.
- Valoración provisional de méritos del concurso.
- Listado definitivo.
- Resolución definitiva.
Antimicrob Agents Chemother. 2013 Jun;57(6):2651-3
Rodríguez-Beltrán J, Rodríguez-Rojas A, Yubero E, Blázquez J.
 Animal fodder is routinely complemented with antibiotics together with other food supplements to improve growth. For instance, sepiolite is currently used as a dietary coadjuvant in animal feed, as it increases animal growth parameters and improves meat and derived final product quality. This type of food additive has so far been considered innocuous for the development and spread of antibiotic resistance.
Animal fodder is routinely complemented with antibiotics together with other food supplements to improve growth. For instance, sepiolite is currently used as a dietary coadjuvant in animal feed, as it increases animal growth parameters and improves meat and derived final product quality. This type of food additive has so far been considered innocuous for the development and spread of antibiotic resistance.
In this study, we demonstrate that sepiolite promotes the direct horizontal transfer of antibiotic resistance plasmids between bacterial species. The conditions needed for plasmid transfer (sepiolite and friction forces) occur in the digestive tracts of farm animals, which routinely receive sepiolite as a food additive. Furthermore, this effect may be aggravated by the use of antibiotics supplied as growth promoters.
Casado-Vela J, Matthiesen R, Sellés S, Naranjo JR.
 Understanding protein interaction networks and their dynamic changes is a major challenge in modern biology. Currently, several experimental and in silico approaches allow the screening of protein interactors in a large-scale manner. Therefore, the bulk of information on protein interactions deposited in databases and peer-reviewed published literature is constantly growing. Multiple databases interfaced from user-friendly web tools recently emerged to facilitate the task of protein interaction data retrieval and data integration. Nevertheless, as we evidence in this report, despite the current efforts towards data integration, the quality of the information on protein interactions retrieved by in silico approaches is frequently incomplete and may even list false interactions. Here we point to some obstacles precluding confident data integration, with special emphasis on protein interactions, which include gene acronym redundancies and protein synonyms.
Understanding protein interaction networks and their dynamic changes is a major challenge in modern biology. Currently, several experimental and in silico approaches allow the screening of protein interactors in a large-scale manner. Therefore, the bulk of information on protein interactions deposited in databases and peer-reviewed published literature is constantly growing. Multiple databases interfaced from user-friendly web tools recently emerged to facilitate the task of protein interaction data retrieval and data integration. Nevertheless, as we evidence in this report, despite the current efforts towards data integration, the quality of the information on protein interactions retrieved by in silico approaches is frequently incomplete and may even list false interactions. Here we point to some obstacles precluding confident data integration, with special emphasis on protein interactions, which include gene acronym redundancies and protein synonyms.
Three human proteins (choline kinase, PPIase and uromodulin) and three different web-based data search engines focused on protein interaction data retrieval (PSICQUIC, DASMI and BIPS) were used to explain the potential occurrence of undesired errors that should be considered by researchers in the field. We demonstrate that, despite the recent initiatives towards data standardization, manual curation of protein interaction networks based on literature searches are still required to remove potential false positives. A three-step workflow consisting of:
(i) data retrieval from multiple databases,
(ii) peer-reviewed literature searches, and
(iii) data curation and integration, is proposed as the best strategy to gather updated information on protein interactions.
Finally, this strategy was applied to compile bona fide information on human DREAM protein interactome, which constitutes liable training datasets that can be used to improve computational predictions.
J Immunol. 2013 Jun 15;190(12):6135-44
Fernández D, Ortiz M, Rodríguez L, García A, Martinez D, Moreno de Alborán I.
 The immune response involves the generation of Ab-secreting cells and memory B cells through a process called terminal B lymphocyte differentiation. This program requires the transcriptional repressor Blimp-1, which inhibits c-myc expression and terminates proliferation. Although the role of c-Myc in cell proliferation is well characterized, it is not known whether it has other functions in terminal differentiation.
The immune response involves the generation of Ab-secreting cells and memory B cells through a process called terminal B lymphocyte differentiation. This program requires the transcriptional repressor Blimp-1, which inhibits c-myc expression and terminates proliferation. Although the role of c-Myc in cell proliferation is well characterized, it is not known whether it has other functions in terminal differentiation.
In this study, we show that c-Myc not only regulates cell proliferation, but it is also essential for Ab-secreting cell function and differentiation in vivo. c-Myc–deficient B lymphocytes hypersecrete IgM and do not undergo Ig class switch recombination (CSR). CSR has been previously linked to proliferation, and in this study we mechanistically link class switching and proliferation via c-Myc. We observed that c-Myc regulates CSR by transcriptionally activating the B cell–specific factor activation-induced cytidine deaminase. By linking cell proliferation and CSR, c-Myc is thus a critical component for a potent immune response.
J Virol. 2013 Apr;87(8):4534-44
Alfonso R, Rodriguez A, Rodriguez P, Lutz T, Nieto A.
 The influenza virus polymerase associates to an important number of transcription-related proteins, including the largest subunit of the RNA polymerase II complex (RNAP II). Despite this association, degradation of the RNAP II takes place in the infected cells once viral transcription is completed. We have previously shown that the chromatin remodeler CHD6 protein interacts with the influenza virus polymerase complex, represses viral replication, and relocalizes to inactive chromatin during influenza virus infection.
The influenza virus polymerase associates to an important number of transcription-related proteins, including the largest subunit of the RNA polymerase II complex (RNAP II). Despite this association, degradation of the RNAP II takes place in the infected cells once viral transcription is completed. We have previously shown that the chromatin remodeler CHD6 protein interacts with the influenza virus polymerase complex, represses viral replication, and relocalizes to inactive chromatin during influenza virus infection.
In this paper, we report that CHD6 acts as a negative modulator of the influenza virus polymerase activity and is also subjected to degradation through a process that includes the following characteristics:
(i) the cellular proteasome is not implicated,
(ii) the sole expression of the three viral polymerase subunits from its cloned cDNAs is sufficient to induce proteolysis, and
(iii) degradation is also observed in vivo in lungs of infected mice and correlates with the increase of viral titers in the lungs.
Collectively, the data indicate that CHD6 degradation is a general effect exerted by influenza A viruses and suggest that this viral repressor may play an important inhibitory role since degradation and accumulation into inactive chromatin occur during the infection.
Cuellar J, Perales-Calvo J, Muga A, Valpuesta JM, Moro F.
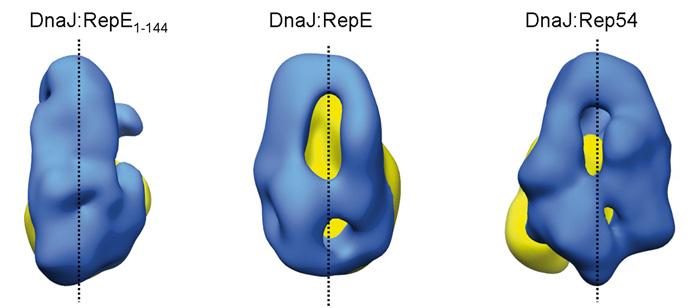 Hsp40 chaperones bind and transfer substrate proteins to Hsp70s and regulate their ATPase activity. The interaction of Hsp40s with native proteins modifies their structure and function. A good model for this function is DnaJ, the bacterial Hsp40 that interacts with RepE, the repressor/activator of plasmid F replication, and together with DnaK regulates its function. We characterize here the structure of the DnaJ:RepE complex by electron microscopy, the first described structure of a complex between an Hsp40 and a client protein. The comparison of the complexes of DnaJ with two RepE mutants reveals an intrinsic plasticity of the DnaJ dimer that allows the chaperone to adapt to different substrates. We also show that DnaJ induces conformational changes in dimeric RepE, which increase the intermonomeric distance and remodel both RepE domains enhancing its affinity for DNA.
Hsp40 chaperones bind and transfer substrate proteins to Hsp70s and regulate their ATPase activity. The interaction of Hsp40s with native proteins modifies their structure and function. A good model for this function is DnaJ, the bacterial Hsp40 that interacts with RepE, the repressor/activator of plasmid F replication, and together with DnaK regulates its function. We characterize here the structure of the DnaJ:RepE complex by electron microscopy, the first described structure of a complex between an Hsp40 and a client protein. The comparison of the complexes of DnaJ with two RepE mutants reveals an intrinsic plasticity of the DnaJ dimer that allows the chaperone to adapt to different substrates. We also show that DnaJ induces conformational changes in dimeric RepE, which increase the intermonomeric distance and remodel both RepE domains enhancing its affinity for DNA.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.





