Natale P, Pazos M, Vicente M.
 Septation in Escherichia coli involves complex molecular mechanisms that contribute to the accuracy of bacterial division. The proto-ring, a complex made up by the FtsZ, FtsA and ZipA proteins, forms at the beginning of the process and directs the assembly of the full divisome. Central to this complex is the FtsZ protein, a GTPase able to assemble into a ring-like structure that responds to several modulatory inputs including mechanisms to position the septum at midcell.
Septation in Escherichia coli involves complex molecular mechanisms that contribute to the accuracy of bacterial division. The proto-ring, a complex made up by the FtsZ, FtsA and ZipA proteins, forms at the beginning of the process and directs the assembly of the full divisome. Central to this complex is the FtsZ protein, a GTPase able to assemble into a ring-like structure that responds to several modulatory inputs including mechanisms to position the septum at midcell.
The connection with the cell wall synthesising machinery stabilizes the constriction of the cytoplasmic membrane. Although a substantial amount of evidence supports this description, many details on how individual divisome elements are structured or how they function are subjected to controversial interpretations. We discuss these discrepancies arising from incomplete data and from technical difficulties imposed by the small size of bacteria. Future work, including more powerful imaging and reconstruction technologies, will help to clarify the missing details on the architecture and function of the bacterial division machinery.
Virology. 2013 Aug 1;442(2):122-31
Pérez Jde J, Udeshi ND, Shabanowitz J, Ciordia S, Juárez S, Scott CL, Olszewski NE, Hunt DF, García JA.
 O-GlcNAcylation is a dynamic protein modification which has been studied mainly in metazoans. We reported previously that an Arabidopsis thaliana O-GlcNAc transferase modifies at least two threonine residues of the Plum pox virus (PPV) capsid protein (CP).
O-GlcNAcylation is a dynamic protein modification which has been studied mainly in metazoans. We reported previously that an Arabidopsis thaliana O-GlcNAc transferase modifies at least two threonine residues of the Plum pox virus (PPV) capsid protein (CP).
Now, six additional residues were shown to be involved in O-GlcNAc modification of PPV CP. CP O-GlcNAcylation was abolished in the PPV CP7-T/A mutant, in which seven threonines were mutated. PPV CP7-T/A infected Nicotiana clevelandii, Nicotiana benthamiana, and Prunus persica without noticeable defects. However, defects in infection of A. thaliana were readily apparent. In mixed infections of wild-type arabidopsis, the CP7-T/A mutant was outcompeted by wild-type virus. These results indicate that CP O-GlcNAcylation has a major role in the infection process. O-GlcNAc modification may have a role in virion assembly and/or stability as the CP of PPV CP7-T/A was more sensitive to protease digestion than that of the wild-type virus.
Environ Microbiol. 2013 Jul 25. doi: 10.1111/1462-2920.12224
Nikel PI, Kim J, de Lorenzo V.
 While the natural niches of the soil bacterium Pseudomonas putida are unlikely to include significant amounts of free glycerol as a growth substrate, this bacterium is genetically equipped with the functions required for its metabolism. We have resorted to deep sequencing of the transcripts in glycerol-grown P. putida KT2440 cells to gain an insight into the biochemical and regulatory components involved in the shift between customary C sources (e.g. glucose or succinate) to the polyol.
While the natural niches of the soil bacterium Pseudomonas putida are unlikely to include significant amounts of free glycerol as a growth substrate, this bacterium is genetically equipped with the functions required for its metabolism. We have resorted to deep sequencing of the transcripts in glycerol-grown P. putida KT2440 cells to gain an insight into the biochemical and regulatory components involved in the shift between customary C sources (e.g. glucose or succinate) to the polyol.
Transcriptomic results were contrasted with key enzymatic activities under the same culture conditions. Cognate expression profiles revealed that genes encoding enzymes of the Entner–Doudoroff route and other catabolic pathways, e.g. the gluconate and 2-ketogluconate loops, were significantly downregulated on glycerol. Yet, the compound simultaneously elicited a gluconeogenic response that indicated an efficient channelling of C skeletons back to biomass build-up through the glyoxylate shunt rather than energization of the cells through downwards pathways, i.e. tricarboxylic acid cycle and oxidative phosphorylation. The simultaneous glycolytic and gluconeogenic metabolic regimes on glycerol, paradoxical as they seem, make sense from an ecological point of view by favouring prevalence versus exploration. This metabolic situation was accompanied by a considerably low expression of stress markers as compared with other C sources.
J Am Chem Soc. 2013 Aug 21;135(33):12366-76
Blanco B, Prado V, Lence E, Otero JM, Garcia-Doval C, van Raaij MJ, Llamas-Saiz AL, Lamb H, Hawkins AR, González-Bello C.
 Shikimate kinase (SK) is an essential enzyme in several pathogenic bacteria and does not have any counterpart in human cells, thus making it an attractive target for the development of new antibiotics. The key interactions of the substrate and product binding and the enzyme movements that are essential for catalytic turnover of the Mycobacterium tuberculosis shikimate kinase enzyme (Mt-SK) have been investigated by structural and computational studies. Based on these studies several substrate analogs were designed and assayed.
Shikimate kinase (SK) is an essential enzyme in several pathogenic bacteria and does not have any counterpart in human cells, thus making it an attractive target for the development of new antibiotics. The key interactions of the substrate and product binding and the enzyme movements that are essential for catalytic turnover of the Mycobacterium tuberculosis shikimate kinase enzyme (Mt-SK) have been investigated by structural and computational studies. Based on these studies several substrate analogs were designed and assayed.
The crystal structure of Mt-SK in complex with ADP and one of the most potent inhibitors has been solved at 2.15 Å. These studies reveal that the fixation of the diaxial conformation of the C4 and C5 hydroxyl groups recognized by the enzyme or the replacement of the C3 hydroxyl group in the natural substrate by an amino group is a promising strategy for inhibition because it causes a dramatic reduction of the flexibility of the LID and shikimic acid binding domains. Molecular dynamics simulation studies showed that the product is expelled from the active site by three arginines (Arg117, Arg136, and Arg58). This finding represents a previously unknown key role of these conserved residues. These studies highlight the key role of the shikimic acid binding domain in the catalysis and provide guidance for future inhibitor designs.
El próximo 4 de octubre de 2013 comienza el XVIII Ciclo de Seminarios del CNB con una conferencia impartida por Matteo Iannacone.
Con la colaboración de Sigma-Aldrich, AppliChem, VWR y Panreac, a lo largo del curso 2013-2014 contaremos con la presencia de 13 magníficos científicos que explicarán sus últimas investigaciones en el campo de la Ciencias de la vida.
A execpción de la ponencia de Philip Cohen que será un jueves, el resto tendrá lugar los viernes a las 12:00 en el Salón de Actos del Centro Nacional de Biotecnología del CSIC.
PLoS One. 2013 Aug 7;8(8):e70364
Banon-Rodriguez I, Saez de Guinoa J, Bernardini A, Ragazzini C, Fernandez E, Carrasco YR, Jones GE, Wandosell F, Anton IM.
The spatial distribution of signals downstream from receptor tyrosine kinases (RTKs) or G-protein coupled receptors (GPCR) regulates fundamental cellular processes that control cell migration and growth. Both pathways rely significantly on actin cytoskeleton reorganization mediated by nucleation-promoting factors such as the WASP-(Wiskott-Aldrich Syndrome Protein) family. WIP (WASP Interacting Protein) is essential for the formation of a class of polarised actin microdomain, namely dorsal ruffles, downstream of the RTK for PDGF (platelet-derived growth factor) but the underlying mechanism is poorly understood.
Using lentivirally-reconstituted WIP-deficient murine fibroblasts we define the requirement for WIP interaction with N-WASP (neural WASP) and Nck for efficient dorsal ruffle formation and of WIP-Nck binding for fibroblast chemotaxis towards PDGF-AA. The formation of both circular dorsal ruffles in PDGF-AA-stimulated primary fibroblasts and lamellipodia in CXCL13-treated B lymphocytes are also compromised by WIP-deficiency. We provide data to show that a WIP-Nck signalling complex interacts with RTK to promote polarised actin remodelling in fibroblasts and provide the first evidence for WIP involvement in the control of migratory persistence in both mesenchymal (fibroblast) and amoeboid (B lymphocytes) motility.
Las células "madre" embrionarias reciben este nombre por su capacidad de generar cualquier tipo celular de las tres capas del embrión a partir de las cuales se originarán los tejidos y órganos en el individuo adulto: el endodermo, el mesodermo o el ectodermo (diferenciación). Además, y al contrario que las células adultas especializadas, las células madre tienen la asombrosa capacidad de poder multiplicarse indefinidamente en el laboratorio (división), lo que las hace tremendamente útiles en el campo de la medicina regenerativa.
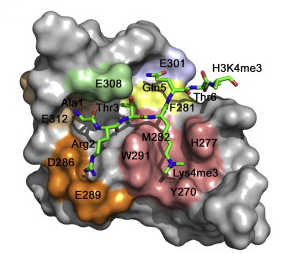 En la actualidad, los científicos tratan de comprender los mecanismos moleculares que regulan en las células madre el equilibrio entre ambos procesos, la división y la diferenciación. Últimamente se han identificado genes que permiten reprogramar células especializadas y convertirlas en células madre pero aun hay muchos genes específicos de estas células cuya función es desconocida.
En la actualidad, los científicos tratan de comprender los mecanismos moleculares que regulan en las células madre el equilibrio entre ambos procesos, la división y la diferenciación. Últimamente se han identificado genes que permiten reprogramar células especializadas y convertirlas en células madre pero aun hay muchos genes específicos de estas células cuya función es desconocida.
Un ejemplo es el gen Dido al que dedica sus esfuerzos el grupo del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Carlos Martínez-A. Este gen aparece evolutivamente en vertebrados y se manifiesta en tres proteínas distintas: Dido1, Dido2 y Dido3. Esta última se expresa en células madre y se modula su expresión en las células especializadas. Se une a estructuras esenciales para la división celular como son el centrosoma y el complejo sinaptonémico. Alteraciones de esta proteína causan síndromes preleucémicos y tumorales y bloquean la capacidad de diferenciarse de las células madre en cualquier tipo celular específico (diferenciación) manteniendo su capacidad de división (proliferación).
En colaboración con investigadores de la Universidad de Colorado, los científicos del CNB acaban de publicar un trabajo en el que identifican al dominio PH de Dido3, y determinan su estructura cristalina a 1.8 A de resolución, como el responsable de la unión al cromosoma. Esta unión causaría modificaciones en la expresión de diferentes genes que le dan a las células madre su capacidad de dividirse y diferenciarse en células adultas.
Además de identificar la zona de la proteína que se une a las histonas que organizan los cromosomas, los investigadores han profundizado en cómo se regula su función según el momento del ciclo celular en que se encuentre la célula. Sus datos indican que cuando las células madre se diferencian, Dido3 es desplazada del cromosoma por el aumento de Dido1. Esto conlleva una disminución en la expresión de genes que dotan a las células madre de su pluripotencia.
En este mismo aspecto, durante la mitosis han observado que Dido3 pasa de estar unido a la cromatina a desplazarse hasta el huso mitótico que dispone a los cromosomas en el ecuador de la célula como paso previo a separarlos hacia los polos cuando se va a producir la división de una célula en dos. De este modo, queda descrita por primera vez a través de Dido relación entre la expresión de genes durante el desarrollo embrionario y la regulación del ciclo celular
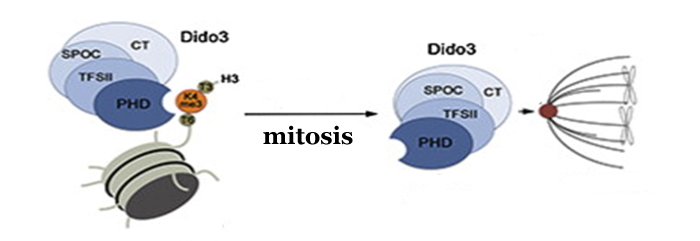
- Gatchalian J, Fütterer A, Rothbart SB, Tong Q, Rincon-Arano H, Sánchez de Diego A, Groudine M, Strahl BD, Martínez-A C, van Wely KH, Kutateladze TG. Dido3 PHD modulates cell differentiation and division. Cell Rep 2013 Jul 2. pii: S2211-1247(13)00292-1.
Environ Microbiol. 2013 Aug 1. doi: 10.1111/1462-2920.12225
Pazos M, Natale P, Margolin W, Vicente M.
 We used bimolecular fluorescence complementation (BiFC) assays to detect protein-protein interactions of all possible pairs of the essential Escherichia coli proto-ring components, FtsZ, FtsA and ZipA, as well as the non-essential FtsZ-associated proteins ZapA and ZapB. We found an unexpected interaction between ZipA and ZapB at potential cell division sites, and when co-overproduced, they induced long narrow constrictions at division sites that were dependent on FtsZ.
We used bimolecular fluorescence complementation (BiFC) assays to detect protein-protein interactions of all possible pairs of the essential Escherichia coli proto-ring components, FtsZ, FtsA and ZipA, as well as the non-essential FtsZ-associated proteins ZapA and ZapB. We found an unexpected interaction between ZipA and ZapB at potential cell division sites, and when co-overproduced, they induced long narrow constrictions at division sites that were dependent on FtsZ.
These assays also uncovered an interaction between ZipA and ZapA that was mediated by FtsZ. BiFC with ZapA and ZapB showed that in addition to their expected interaction at midcell, they also interact at the cell poles. BiFC detected interaction between FtsZ and ZapB at midcell and close to the poles. Results from the remaining pairwise combinations confirmed known interactions between FtsZ and ZipA, and ZapB with itself.
J Biol Chem. 2013 Jun 7;288(23):16998-7007
Daudén MI, Martín-Benito J, Sánchez-Ferrero JC, Pulido-Cid M, Valpuesta JM, Carrascosa JL.
 During bacteriophage morphogenesis DNA is translocated into a preformed prohead by the complex formed by the portal protein, or connector, plus the terminase, which are located at an especial prohead vertex. The terminase is a powerful motor that converts ATP hydrolysis into mechanical movement of the DNA. Here, we have determined the structure of the T7 large terminase by electron microscopy.
During bacteriophage morphogenesis DNA is translocated into a preformed prohead by the complex formed by the portal protein, or connector, plus the terminase, which are located at an especial prohead vertex. The terminase is a powerful motor that converts ATP hydrolysis into mechanical movement of the DNA. Here, we have determined the structure of the T7 large terminase by electron microscopy.
The five terminase subunits assemble in a toroid that encloses a channel wide enough to accommodate dsDNA. The structure of the complete connector-terminase complex is also reported, revealing the coupling between the terminase and the connector forming a continuous channel. The structure of the terminase assembled into the complex showed a different conformation when compared with the isolated terminase pentamer. To understand in molecular terms the terminase morphological change, we generated the terminase atomic model based on the crystallographic structure of its phage T4 counterpart. The docking of the threaded model in both terminase conformations showed that the transition between the two states can be achieved by rigid body subunit rotation in the pentameric assembly. The existence of two terminase conformations and its possible relation to the sequential DNA translocation may shed light into the molecular bases of the packaging mechanism of bacteriophage T7.
J Struct Biol. 2013 Aug 6. pii: S1047-8477(13)00195-0
Vargas J, Abrishami V, Marabini R, de la Rosa-Trevín JM, Zaldivar A, Carazo JM, Sorzano CO.
 Three-dimensional reconstruction of biological specimens using electron microscopy by single particle methodologies requires the identification and extraction of the imaged particles from the acquired micrographs. Automatic and semiautomatic particle selection approaches can localize these particles, minimizing the user interaction, but at the cost of selecting a non-negligible number of incorrect particles, which can corrupt the final three-dimensional reconstruction.
Three-dimensional reconstruction of biological specimens using electron microscopy by single particle methodologies requires the identification and extraction of the imaged particles from the acquired micrographs. Automatic and semiautomatic particle selection approaches can localize these particles, minimizing the user interaction, but at the cost of selecting a non-negligible number of incorrect particles, which can corrupt the final three-dimensional reconstruction.
In this work, we present a novel particle quality assessment and sorting method that can separate most erroneously picked particles from correct ones. The proposed method is based on multivariate statistical analysis of a particle set that has been picked previously using any automatic or manual approach. The new method uses different sets of particle descriptors, which are morphology-based, histogram-based and signal to noise analysis based. We have tested our proposed algorithm with experimental data obtaining very satisfactory results. The algorithm is freely available as a part of the Xmipp 3.0 package.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.






