Hernando-Pérez M, Martín-González N, Pérez-Illana M, Suomalainen M, Condezo GN, Philomena Ostapchuk P, Gallardo J, Menéndez M, Greber UF, Hearing P, de Pablo PJ, San Martín C.
Abstract
Adenovirus minor coat protein VI contains a membrane-disrupting peptide that is inactive when VI is bound to hexon trimers. Protein VI must be released during entry to ensure endosome escape. Hexon:VI stoichiometry has been uncertain, and only fragments of VI have been identified in the virion structure. Recent findings suggest an unexpected relationship between VI and the major core protein, VII. According to the high-resolution structure of the mature virion, VI and VII may compete for the same binding site in hexon; and noninfectious human adenovirus type 5 particles assembled in the absence of VII (Ad5-VII-) are deficient in proteolytic maturation of protein VI and endosome escape. Here we show that Ad5-VII- particles are trapped in the endosome because they fail to increase VI exposure during entry. This failure was not due to increased particle stability, because capsid disruption happened at lower thermal or mechanical stress in Ad5-VII- compared to wild-type (Ad5-wt) particles. Cryoelectron microscopy difference maps indicated that VII can occupy the same binding pocket as VI in all hexon monomers, strongly arguing for binding competition. In the Ad5-VII- map, density corresponding to the immature amino-terminal region of VI indicates that in the absence of VII the lytic peptide is trapped inside the hexon cavity, and clarifies the hexon:VI stoichiometry conundrum. We propose a model where dynamic competition between proteins VI and VII for hexon binding facilitates the complete maturation of VI, and is responsible for releasing the lytic protein from the hexon cavity during entry and stepwise uncoating.
- Los dos proyectos que se desarrollan en el CNB están progresando muy rápido, todo lo técnicamente posible, y cuentan con los recursos necesarios gracias a la financiación del Gobierno
- El proyecto de Enjuanes y Sola ha conseguido un ADN complementario completo del coronavirus SARS-CoV-2 y en paralelo se han preparado los test en modelos animales
- El proyecto de Mariano Esteban cuenta ya con un candidato a vacuna y se han vacunado ratones para probar la respuesta inmune protectiva frente al coronavirus
Abierto el plazo de solicitud para la nueva convocatoria de becas de colaboración de introducción a la investigación JAE Intro SOMdM 2020 orientadas a estudiantes universitarios de máster en alguno de los Centros de Excelencia Severo Ochoa y Unidades de Excelencia María de Maeztu del CSIC. El plazo de recepción de solicitudes está abierto hasta el 15 de junio de 2020.
Esta convocatoria está dirigida a estudiantes que vayan a cursar un Máster Universitario durante el curso 2020/2021. Los solicitantes deberán haber finalizado la licenciatura o grado en el curso académico 2018/2019 o estar finalizando sus estudios en el curso académico 2019-2020, (consulten las excepciones en las bases completas) y acreditar una nota media igual o superior a 8,00.
Las bases completas de la convocatoria se pueden consultar aquí
NUEVO: 23 de JUNIO: lista provisional de solicitantes admitidos y excluidos
NUEVO: 9 de JULIO: lista definitiva de solicitantes admitidos y excluidos
NUEVO: 22 de JULIO: CONCESIÓN
El CNB-CSIC ofrecerá hasta 6 becas, una por departamento y cada una contará con una dotación total de 7200 euros en nueve mensualidades. Para más información de los grupos ofertados revisa nuestro listado:
Departamento de Estructura de Macromoléculas
Departamento de Biología Molecular y Celular
Departamento de Biotecnología Microbiana
Departamento de Genética Molecular de Plantas
Departamento de Inmunología y Oncología
Departamento de Biología de Sistemas
El Centro Nacional de Biotecnología es un centro de investigación multidisciplinar que cuenta con grupos científicos dedicados a la virología y la inmunología desde diferentes aproximaciones científicas. Desde el conocimiento de los aspectos básicos de mecanismos biológicos y estructurales de infecciones virales a la respuesta inmune que se produce en el organismo infectado, estos grupos han unido sus esfuerzos y desarrollan diferentes estrategias para ofrecer respuestas a la actual pandemia de COVID-19 causada por el virus SARS-CoV-2. Nuestras líneas de actuación comprenden más de quince proyectos liderados por investigadores e investigadoras del centro.
Estos proyectos se encuentran incluidos en la Plataforma Temática Interdisciplinar (PTI) denominada Salud Global/Global Health del CSIC, en la que colaboran más de 200 grupos de investigación de diferentes especialidades, para abordar los retos que plantea la epidemia del coronavirus desde el punto de vista de la Ciencia, con el objetivo de plantear soluciones a corto, medio y sobre todo largo plazo.
 Desarrollo de una vacuna contra el SARS-CoV-2 basada en replicones no infectivos
Desarrollo de una vacuna contra el SARS-CoV-2 basada en replicones no infectivos
Investigadores principales: Luis Enjuanes, Isabel Sola, Sonia Zuñiga
El objetivo de este proyecto es generar el virus SARS-CoV-2 en el laboratorio mediante el ensamblaje de fragmentos de ADN sintético y eliminar de su genoma los genes responsables de la virulencia con técnicas de genética inversa para introducir mutaciones atenuantes y obtener derivados no infectivos y altamente inmunogénicos como candidatos a vacunas contra el SARS-CoV-2. En paralelo, se está trabajando en el desarrollo de modelos animales (ratones transgénicos) para la validación de vacunas y otros agentes terapéuticos contra COVID-19.
 Desarrollo de una vacuna contra el SARS-CoV-2 basada en el vector MVA no-replicativo expresando diversos antígenos virales
Desarrollo de una vacuna contra el SARS-CoV-2 basada en el vector MVA no-replicativo expresando diversos antígenos virales
Investigadores principales: Mariano Esteban, Juan García Arriaza, Carmen E. Gómez
Este proyecto se basa en la generación de vectores virales del virus Vaccinia (MVA) que contengan secuencias codificantes de alguna de las proteínas inmunogénicas de la superficie del SARS-CoV-2. Ya que esta aproximación no requiere de la utilización del virus SARS-CoV-2 completo, permite trabajar en condiciones de menor nivel de seguridad biológica. Debido a su alta atenuación, ya demostrada con vacunas desarrolladas previamente con éxito contra la viruela, la vacuna MVA-COVID-19 sería segura y se podría administrar a todo tipo de población, incluyendo personas con inmunodeficiencias.
 Cribado masivo de compuestos antivirales contra el SARS-CoV-2
Cribado masivo de compuestos antivirales contra el SARS-CoV-2
Investigadores principales: Pablo Gastaminza, Urtzi Garaigorta
Plataforma para el cribado masivo (screening) de compuestos antivirales, mediante dos fases, una que se desarrolla en laboratorio P2 con un modelo de coronavirus del catarro común y otra que se realiza en laboratorio P3 donde los compuestos seleccionados se evalúan contra SARS-CoV2. Se estudian fármacos de reposicionamiento que ya se están utilizando en clínica y que por lo tanto supondrían una manera rápida de encontrar tratamientos eficaces en humanos y también librerías de compuestos, en colaboración con otros proyectos liderados por grupos CSIC, para identificar nuevos compuestos y analizar su mecanismo de acción antiviral. En colaboración con otros proyectos liderados por grupos CSIC se está llevando a cabo el cribado de diferentes colecciones de moléculas que está permitiendo abordar tanto el reposicionamiento de compuestos de uso clínico como la exploración de nuevos compuestos antivirales.
 Validación preclínica de agentes terapéuticos contra el SARS-CoV-2
Validación preclínica de agentes terapéuticos contra el SARS-CoV-2
Investigadores principales: Luis Enjuanes, Isabel Sola, Sonia Zuñiga, Leonor Kremer
Este grupo de investigación dispone de diferentes modelos in vitro e in vivo para el estudio de la patogénesis de coronavirus humanos y está finalizando el desarrollo de modelos específicos para el SARS-CoV-2. Estos modelos se utilizarán para la validación preclínica de compuestos terapéuticos contra el SARS-CoV-2, incluyendo desarrollos propios y proyectos en colaboración con la industria farmacéutica española para el reposicionamiento de fármacos y estrategias de inmunoterapia.
 Modelos computacionales para el diseño de intervenciones terapéuticas contra el SARS-CoV-2
Modelos computacionales para el diseño de intervenciones terapéuticas contra el SARS-CoV-2
Investigadores principales: José Ramón Valverde, Luis Enjuanes, J. Manuel Honrubia
Esta línea de actuación aplica métodos computacionales a varios aspectos claves en la investigación sobre el SARS-CoV-2, incluyendo la modelización de mecanismos por los que el virus podría desencadenar los diferentes síntomas de COVID-19, el análisis de la interacción de anticuerpos con el SARS-CoV-2 para el diseño de anticuerpos neutralizantes, así como estudios in silico de la dinámica de interacción entre proteínas de superficie del virus y su receptor con el propósito de generar un modelo de ratón humanizado para estudios pre-clínicos.
 Desarrollo de anticuerpos pequeños (nanobodies) frente al SARS-CoV-2
Desarrollo de anticuerpos pequeños (nanobodies) frente al SARS-CoV-2
Investigadores principales: Luis Ángel Fernández, José María Casasnovas, Víctor de Lorenzo
El objetivo de esta línea de actuación es la obtención de anticuerpos neutralizantes frente a SARS-CoV-2, utilizando la tecnología de generación de anticuerpos de dominio simple con una región VH humana y un dominio VHH de camélido (nanobodies) en bacterias, partiendo de librerías de nanobodies disponibles, librerías que se están generando mediante inmunización de dromedarios con antígenos del SARS-CoV-2, colecciones de anticuerpos humanos, o mediante síntesis y mutagénesis de anticuerpos neutralizantes.
 Nanopartículas supresoras con tropismo a pulmón para frenar la tormenta de citoquinas causante de la inmunopatología pulmonar y la replicación viral.
Nanopartículas supresoras con tropismo a pulmón para frenar la tormenta de citoquinas causante de la inmunopatología pulmonar y la replicación viral.
Investigador principal: Domingo F. Barber
Diseño de nanopartículas que puedan usarse para reducir la inflamación que se produce, especialmente en los pulmones, al infectarse con SARS-CoV-2. El proyecto permitirá entender como interfieren en la reproducción del virus las nanopartículas de óxido de hierro (NPOH), las cuales normalmente se usan en resonancias magnética o para el tratamiento de anemias (FeraSpin™y Feraheme), así como diseñar NPOH que puedan emplearse para reducir la inflamación pulmonar generada en algunos casos de infección por SARS2. Estas nanopartículas podrían también se utilizadas para mitigar la inflamación causada por un alto número de enfermedades inflamatorias.
 Destrucción del genoma ARN del coronavirus mediante CRIPR-Cas13d.
Destrucción del genoma ARN del coronavirus mediante CRIPR-Cas13d.
Investigadora principal: Dolores Rodríguez
Este proyecto plantea usar una nueva variante de las herramientas CRISPR de edición genética, la proteína Cas13d, con actividad RNAsa específica dirigida por una guía de RNA, para destruir el genoma RNA del coronavirus dentro de las células. La no toxicidad y eficiencia de los reactivos CRISPR se validará in vivo en embriones de pez cebra. La eficacia del proyecto se explorará con ayuda de dos virus relacionados con el coronavirus que permiten trabajar en condiciones BSL2, antes de adaptar el proyecto para el coronavirus SARS-CoV-2.
 Ensayos para la identificación de individuos seropositivos para COVID-19 mediante antígenos recombinantes producidos en diferentes sistemas de expresión (bacterias, células de insecto y mamífero).
Ensayos para la identificación de individuos seropositivos para COVID-19 mediante antígenos recombinantes producidos en diferentes sistemas de expresión (bacterias, células de insecto y mamífero).
Investigadores principales: Francisco Rodríguez, Hugh Reyburn, José María Casasnovas, Mar Valés, José Miguel Rodríguez Frade
Gracias a la colaboración entre varios grupos de investigación, ha sido posible poner a punto la producción de distintas proteínas del SARS-CoV-2 y su incorporación a test de diagnóstico de seropositividad. La capacidad de estos antígenos para detectar la presencia de anticuerpos frente a SARS-CoV-2 en sueros de pacientes convalecientes abre el camino hacia el desarrollo de pruebas diagnósticas para identificar diferentes estadíos de inmunidad en personas infectadas por SARS-CoV-2, herramienta clave para estudios epidemiológicos, detectar la presencia de anticuerpos neutralizantes o hacer seguimiento temporal de la inmunidad de los pacientes. Aspectos muy importantes en los procesos de desescalada y vuelta a la rutina.
 Caracterización de la respuesta inmunológica al virus SARS-COV-2 para el control de la fase epidémica
Caracterización de la respuesta inmunológica al virus SARS-COV-2 para el control de la fase epidémica
Investigadores principales: Margarita del Val (CBMSO) e Isabel Mérida
A corto plazo este proyecto determinará el porcentaje de una población homogénea, en concreto niños de entre 5-16 años que estarán inmunizados frente al coronavirus, lo que permitirá estimar la vulnerabilidad de la población española a una nueva oleada de la infección COVID-19. A medio plazo se conocerá la capacidad que tiene el virus SARS-CoV2 para producir la enfermedad COVID-19 en la población y las patologías que tiene asociadas. Y, en función de esto, guiar terapias para su tratamiento y el desarrollo de vacunas.
 Predicción de la dinámica epidémica de COVID-19
Predicción de la dinámica epidémica de COVID-19
Investigadores principales: Susanna Manrubia, Saúl Ares
El enfoque de este proyecto es el desarrollo de herramientas que permitan gestionar, mediante modelos predictivos, las medidas de distanciamiento social adecuadas para contener o prevenir la expansión del COVID-19. La primera fase del proyecto se centra en el análisis de datos de evolución de la pandemia en España y en la elaboración de modelos predictivos y consensuados. En una segunda fase, se abordará el perfeccionamiento de modelos predictivos y de informes de potencial utilidad ante una próxima pandemia y el estudio de modelos explicativos.
 Desarrollo y validación experimental de sistemas de esterilización y descontaminación para la inactivación del SARS-CoV-2
Desarrollo y validación experimental de sistemas de esterilización y descontaminación para la inactivación del SARS-CoV-2
Investigador principal: Fernando Usera
El laboratorio de nivel de contención biológica 3 (NCB-3) es la infraestructura clave del CNB para la realización de experimentos con el SARS-CoV-2. Además, el laboratorio participa en una serie de estudios en colaboración con entidades públicas y empresas que pretenden desarrollar y validar métodos de esterilización y descontaminación para la inactivación del SARS-CoV-2 en diferentes ambientes y superficies contaminadas.
 Proteómica del SARS-CoV-2
Proteómica del SARS-CoV-2
Investigadores principales: Fernando Corrales, Alberto Paradela
El laboratorio de Proteómica del CNB participa en varios proyectos colaborativos que estudian la interacción del SARS-CoV-2 con el hospedador y la respuesta inmunológica de pacientes de COVID-19 para identificar epítopos virales y perfiles de anticuerpos que permitan elaborar estrategias de vacunación, estratificación, e identificación de individuos protegidos, proporcionando la base de nuevas estrategias de diagnóstico, terapia y vacunación, necesarias para controlar la presente y futuras pandemias. Corto plazo: Desarrollo de moléculas terapéuticas facilitando métodos de control de calidad. Medio: Definir la respuesta inmune humoral de pacientes con COVID-19 (respuesta protectora de nuestro cuerpo frente a partículas extracelulares del virus). Diagnóstico y estratificación de pacientes. Largo: Definir mecanismos asociados al proceso de la infección. Nuevas vías diagnóstico y/o terapia. Forma parte de la iniciativa internacional COVID19 Mass Spectrometry Community.
 Biología estructural del SARS-CoV-2
Biología estructural del SARS-CoV-2
Investigadores principales: José María Valpuesta, José Ruiz Castón, Jaime Martín Benito, Carmen San Martín, Mark van Raaij
Con el objetivo de guiar el diseño de anticuerpos, compuestos antivirales y otras herramientas moleculares para combatir el COVID-19, se están utilizando técnicas de cristalografía de rayos X, criomicroscopía electrónica y criomicroscopía correlativa, para conocer la estructura del virus, de sus componentes y del proceso de infección. Asimismo, se está trabajando en el diseño de pseudopartículas virales quiméricas de regiones antigénicas de SARS-CoV-2 para validar su potencial de generar una respuesta protectora frente al SARS-CoV-2.
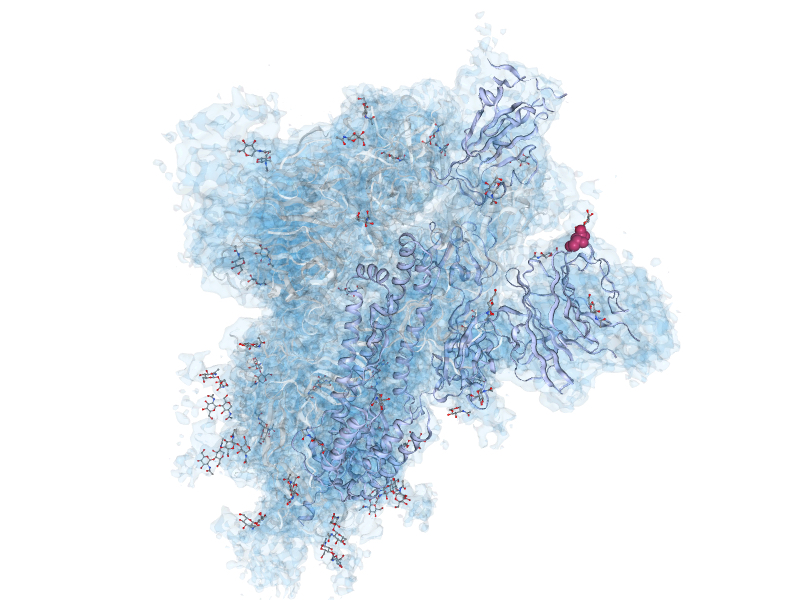 Estudios estructurales de la proteína S
Estudios estructurales de la proteína S
José María Carazo, Carlos Óscar Sánchez-Sorzano
Este proyecto forma parte de una colaboración internacional con el objetivo de determinar la estructura de la glicoproteina S (spike) a alta resolución mediante criomicroscopía electrónica. Los resultados ayudarán a comprender mejor las particularidades de la interacción del SARS-CoV-2 con su receptor lo cual podría explicar su mayor capacidad infectiva. Además, se analizarán imágenes de complejos de spike con anticuerpos humanos neutralizantes, procedentes de pacientes que hayan superado la enfermedad, aportando información relevante para el diseño de estrategias terapéuticas.
 Nodo de información sobre estructura-función del SARS-CoV-2
Nodo de información sobre estructura-función del SARS-CoV-2
José María Carazo, Carlos Óscar Sánchez-Sorzano
El objetivo de este proyecto es integrar y hacer disponible a la comunidad científica en el plazo más corto posible toda la información estructural y biomédica que se está generando en el mundo en torno al SARS-CoV-2. En coordinación con el Instituto Nacional de Bioinformática y las infraestructuras europeas Instruct, ELIXIR y EOSC, se está construyendo un potente nodo de información interactivo en web llamado 3DBionotes con la visión de ser desplegado en un futuro portal COVID19 de la iniciativa European Open Science Cloud (EOSC).
Proyecto de divulgación "Ciencia y Responsabilidad frente a la covid-19", dirigido por Susanna Manrubia y financiado por el programa Cuenta la Ciencia de la Fundación General CSIC ha desarrollado nuevos formatos de difusión de la investigación del CNB como son los comics, y la elaboración un vídeo resumen de los logros del CNBen este tiempo
Debido al interés social de la pandemia causada por el SARS-Co2, el CSIC y el CNB están recibiendo donaciones de instituciones y particulares. Desde aquí mostramos nuestro agradecimiento tanto a las empresas como a los particulares anónimos por su implicación en nuestra investigación.

Nueva convocatoria para solicitar becas JAE de Introducción a la Investigación en centros del CSIC. Las 250 becas ofertadas becas están orientadas a estudiantes universitarios, especialmente aquellos en el último curso de grado universitario y tendrán una duración de 5 meses consecutivos, iniciándose en septiembre u octubre de 2020.
El plazo de presentación de solicitudes se ha vuelto a abrir y termina el 10 de junio (incluido)
El Centro Nacional de Biotecnología recibirá 11 estudiantes en el curso 2020-2021
16 de JULIO 2020: RESOLUCION DEFINITIVA publicada en la página del CSIC
El próximo miercoles 29 de enero el CNB organiza un simposio en honor a Amelia Nieto, investigadora científica que ha pertenecido al CNB desde sus orígenes.
Por su jubilación, el centro reune a algunos investigadores que han salido de su grupo en un simposio realizado en su honor bajo el título "Looking at Cell Biology from a virus perspective". En el evento participarán entre otros Maite Huarte, Tomás Aragón y Puri Fortes del Centro de Investigaciones Médicas Aplicadas (CIMA) de Pamplona; Juan Varcarcel y Susana de la Luna del Centre Reserca Genòmica (CRG) de Barcelona; desde Suiza vendrán Santiago F. González de la Università della Svizzera en Bellinzona y Emilio Yángüez del Functional Genomic Service del ETH de Zurich.
Este evento cuenta con el apoyo de The Company of Biologist a través de su programa de Becas para la celebración de reuniones científicas.
- Carmen Elena Gómez recibe el Premio de Investigación de la Real Academia Nacional de Farmacia (RANF)
- Mariano Esteban recibe la medalla de oro Carracido por su trayectoria científica y académica.
Carmen Elena Gómez, investigadora del Departamento de Biología Molecular y Celular del CNB ha sido distinguida (ex aequo) con el Premio de Investigación de la Real Academia Nacional de Farmacia (RANF). La entrega de este galardón ha tenido lugar en la sede de la Academia el día 15 de enero de 2020.
Descubre en el campus de Cantoblanco a científicas premiadas y olvidadas por el Nobel
Entre el 3 y el 14 de febrero se realizará un escape road en las facultades de ciencias y biología donde se presentarán perfiles de mujeres galardonadas con el Nobel en un recorrido en el que los visitantes responderán preguntas sobre las investigadoras.
- Los participantes buscarán, a través de una aplicación móvil, carteles ocultos de grandes científicas que no ganaron el Nobel.
- Este “escape road” es una actividad conjunta organizada por los centros del CSIC del campus de Cantoblanco: (Instituto de Ciencia de Materiales de Madrid, Instituto de Cerámica y Vidrio, Instituto de Catálisis y Petroquímica, Centro Nacional de Biotecnología, Centro de Biología Molecular, Instituto de Ciencias Matemáticas, Instituto de Física Teórica e Instituto de Investigación en Ciencias de la Alimentación.)
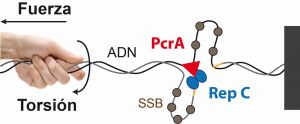
Los plásmidos son moléculas de ADN circulares presentes en bacterias que se replican (duplican) de forma independiente del cromosoma bacteriano. La transmisión de plásmidos entre bacterias puede suponer la ganancia o la perdida de una función determinada en la célula receptora. Por ejemplo, algunos genes contenidos en plásmidos son capaces de conferir resistencia a antibióticos, y la transmisión de estos genes puede ser beneficiosa para la supervivencia bacteriana, ya que se convertirán en resistentes a esos antibióticos.
El sistema más habitual de duplicación de estos plásmidos se llama replicación por circulo rodante (RCR). Este proceso requiere la unión de la proteína Rep a una secuencia determinada del ADN, el corte de una de las cadenas del ADN y la separación de la doble cadena a medida que la helicasa PcrA se mueve por una de ellas. Sin embargo, el mecanismo por el cual se inicia la replicación y se desenrolla la doble hélice del ADN no se conoce con detalle.
Investigadores del Centro Nacional de Biotecnología, en colaboración con un grupo de científicos de la Universidad de Pittsburgh (Estados Unidos) han sido capaces de observar a nivel molecular la interacción de las proteínas Rep y PcrA en el proceso de replicación de plásmidos portadores de genes de resistencia a antibióticos.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.