- El proyecto LIFE LEMNA presenta el primer prototipo semiindustrial de Europa basado en la tecnología de cultivo de lenteja de agua (Lemna) para la recuperación de nutrientes contenidos en los purines excedentarios de porcino.
- La instalación, de 250 m2, puede producir más de 17t de biomasa/ha·año, con una productividad de proteína de origen vegetal 6-7 veces mayor que la obtenida en el cultivo de soja
- La lenteja de agua es una pequeña planta acuática menor de un cm, flotante, de forma circular y de color verde hierba intenso, que es capaz de absorber los nutrientes contenidos en los residuos ganaderos de forma muy eficiente y transformarlos en una biomasa rica en proteínas.
El proyecto europeo LIFE LEMNA busca soluciones que reduzcan el impacto medioambiental generado por la acumulación de nutrientes en los purines excedentarios de la ganadería intensiva. Encontrar soluciones ambiental y económicamente viables para estos materiales es una prioridad en España y el resto de la UE debido a la problemática de contaminación por nitratos y emisiones de CO2 asociada. Además, reducir la huella ambiental de los productos cárnicos va a resultar un factor de competitividad en el futuro por lo que el sector necesita encontrar soluciones.
Para ayudar a resolver este problema, el centro tecnológico AINIA, junto con el Centro Nacional de Biotecnología (CNB) perteneciente al Consejo Superior de Investigaciones Científicas (CSIC) y las empresa Porgaporcs y ECOBIOGAS, han desarrollado la primera planta piloto a escala semi industrial de Europa que permite recuperar los nutrientes (nitrógeno, fósforo) de los purines, y reutilizarlos en la propia zona en la que se generan.
Plant, Cell & Environment 08 July 2019
J Ochoa de Eribe, A Valli, M Martín‐Trillo, C Simón‐Mateo, JA García, B Rodamilans.
Abstract
Plants use RNA silencing as a strong defensive barrier against virus challenges, and viruses counteract this defence by using RNA silencing suppressors (RSSs). With the objective of identifying host factors helping either the plant or the virus in this interaction, we have performed a yeast two hybrid screen using P1b, the RSS protein of the ipomovirus Cucumber vein yellowing virus (CVYV, family Potyviridae), as a bait. The C‐8 sterol isomerase HYDRA1 (HYD1), an enzyme involved in isoprenoid biosynthesis and cell membrane biology, and required for RNA silencing, was isolated in this screen. The interaction between CVYV P1b and HYD1 was confirmed in planta by Bimolecular Fluorescence Complementation assays. We demonstrated that HYD1 enhances RNA silencing activity in the presence of CVYV P1b in an agroinfiltration assay. Moreover, expression of HYD1 inhibited the infection of the potyvirus Plum pox virus, especially when antiviral RNA silencing was boosted by high temperature or by co‐expression of homologous sequences. Our results reinforce previous evidence highlighting the relevance of particular composition and structure of cellular membranes for RNA silencing and viral infection. We report a new interaction of an RSS protein from the Potyviridae family with a member of the isoprenoid biosynthetic pathway.
Viruses undergo mesoscopic morphological changes as they interact with host interfaces and in response to chemical cues. The dynamics of these changes, over the entire temporal range relevant to virus processes, are unclear. Here, we report on creep compliance experiments on a small icosahedral virus under uniaxial constant stress. We find that even at small stresses, well below the yielding point and generally thought to induce a Hookean response, strain continues to develop in time via sparse, randomly distributed, relatively rapid plastic events. The intermittent character of mechanical compliance only appears above a loading threshold, similar to situations encountered in granular flows and the plastic deformation of crystalline solids. The threshold load is much smaller for the empty capsids of the brome mosaic virus than for the wild-type virions. The difference highlights the involvement of RNA in stabilizing the assembly interface. Numerical simulations of spherical crystal deformation suggest intermittency is mediated by lattice defect dynamics and identify the type of compression-induced defect that nucleates the transition to plasticity.
doi: 10.1021/acsnano.9b02133.
- Las chaperonas moleculares son clave para el equilibrio entre la síntesis y degradación de las proteínas celulares
- Para analizar la función de estos complejos y conocer su estructura, los científicos han empleado el criomicroscopio electrónico del Centro Nacional de Biotecnología del CSIC
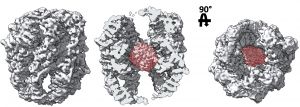
Un trabajo codirigido por investigadores del Centro Nacional de Biotecnología del Consejo Superior de Investigaciones Científicas (CSIC) ha determinado el mecanismo de funcionamiento del complejo CCT, una de las chaperonas moleculares más importantes en organismos eucariotas. Los científicos, que publican sus resultados en el último número de la revista Nature Communications, han empleado un criomicroscopio electrónico de última generación.
Nature Communications 10, Article number: 2865 (2019)
Jorge Cuéllar, W. Grant Ludlam, Nicole C. Tensmeyer, Takuma Aoba, Madhura Dhavale, César Santiago, M. Teresa Bueno-Carrasco, Michael J. Mann, Rebecca L. Plimpton, Aman Makaju, Sarah Franklin, Barry M. Willardson & José M. Valpuesta.
Abstract
The mechanistic target of rapamycin (mTOR) kinase forms two multi-protein signaling complexes, mTORC1 and mTORC2, which are master regulators of cell growth, metabolism, survival and autophagy. Two of the subunits of these complexes are mLST8 and Raptor, β-propeller proteins that stabilize the mTOR kinase and recruit substrates, respectively. Here we report that the eukaryotic chaperonin CCT plays a key role in mTORC assembly and signaling by folding both mLST8 and Raptor. A high resolution (4.0 Å) cryo-EM structure of the human mLST8-CCT intermediate isolated directly from cells shows mLST8 in a near-native state bound to CCT deep within the folding chamber between the two CCT rings, and interacting mainly with the disordered N- and C-termini of specific CCT subunits of both rings. These findings describe a unique function of CCT in mTORC assembly and a distinct binding site in CCT for mLST8, far from those found for similar β-propeller proteins.
Investigadores del CSIC colaboran en un estudio internacional sobre el origen y la evolución de las variedades europeas de la patata, con la ayuda de las colecciones históricas de los herbarios. La adaptación fuera de su región de origen puede haber seguido rutas similares en Europa y el Sur de América.
Plant Cell. 2019 Jun;31(6):1222-1237.
Méndez-Vigo B, Ausín I, Zhu W2, Mollá-Morales A, Balasubramanian S, Alonso-Blanco C.
Abstract
Understanding how plants adapt to ambient temperatures has become a major challenge prompted by global climate change. This has led to the identification of several genes regulating the thermal plasticity of plant growth and flowering time. However, the mechanisms accounting for the natural variation and evolution of such developmental plasticity remain mostly unknown. In this study, we determined that natural variation at ICARUS2 (ICA2), which interacts genetically with its homolog ICA1, alters growth and flowering time plasticity in relation to temperature in Arabidopsis (Arabidopsis thaliana). Transgenic analyses demonstrated multiple functional effects for ICA2 and supported the notion that structural polymorphisms in ICA2 likely underlie its natural variation. Two major ICA2 haplogroups carrying distinct functionally active alleles showed high frequency, strong geographic structure, and significant associations with climatic variables related to annual and daily fluctuations in temperature. Genome analyses across the plant phylogeny indicated that the prevalent plant ICA genes encoding two tRNAHis guanylyl transferase 1 units evolved ∼120 million years ago during the early divergence of mono- and dicotyledonous clades. In addition, ICA1/ICA2 duplication occurred specifically in the Camelineae tribe (Brassicaceae). Thus, ICA2 appears to be ubiquitous across plant evolution and likely contributes to climate adaptation through modifications of thermal developmental plasticity in Arabidopsis.
doi: 10.1105/tpc.18.00938. Epub 2019 Apr 16
- Identificados en ‘Arabidopsis thaliana’ los genes que regulan un nuevo mecanismo de adaptación a altas temperaturas
- La interacción genética entre dos genes duplicados regula el ciclo de la división celular, según un estudio del CSIC
Las plantas están sometidas a fluctuaciones de temperatura diarias y estacionales que provocan cambios en sus patrones de expresión genética. Esta plasticidad les permite adaptarse a las condiciones ambientales mediante cambios en su ritmo de crecimiento y tiempo de floración.
Entender cómo las plantas de diferentes regiones del mundo se adaptan a las temperaturas del ambiente en el que crecen es el objetivo de las investigaciones de un equipo de científicos del Consejo Superior de Investigaciones Científicas (CSIC) en el Centro Nacional de Biotecnología. En su último trabajo, publicado en la revista The Plant Cell, los investigadores han identificado en la planta modelo Arabidopsis thaliana dos genes duplicados, ICARUS1 y 2 (ICA1 e ICA2), que están implicados en la regulación de la respuesta térmica del desarrollo de las plantas.
J Immunother Cancer. 2019 Jun 13;7(1):151.
Ogando J, Sáez ME, Santos J, Nuevo-Tapioles C, Gut M, Esteve-Codina A, Heath S, González-Pérez A, Cuezva JM, Lacalle RA, Mañes S
Abstract
BACKGROUND: Binding of the programmed death-1 (PD-1) receptor to its ligands (PD-L1/2) transduces inhibitory signals that promote exhaustion of activated T cells. Blockade of the PD-1 pathway is widely used for cancer treatment, yet the inhibitory signals transduced by PD-1 in T cells remain elusive.
METHODS: Expression profiles of human CD8+ T cells in resting, activated (CD3 + CD28) and PD-1-stimulated cells (CD3 + CD28 + PD-L1-Fc) conditions were evaluated by RNA-seq. Bioinformatic analyses were used to identify signaling pathways differentially regulated in PD-1-stimulated cells. Metabolic analyses were performed with SeaHorse technology, and mitochondrial ultrastructure was determined by transmission electron microscopy. PD-1-regulated mitochondrial genes were silenced using short-hairpin RNA in primary cells. Blue native gel electrophoresis was used to determine respiratory supercomplex assembly.
RESULTS: PD-1 engagement in human CD8+ T cells triggers a specific, progressive genetic program different from that found in resting cells. Gene ontology identified metabolic processes, including glycolysis and oxidative phosphorylation (OXPHOS), as the main pathways targeted by PD-1. We observed severe functional and structural alterations in the mitochondria of PD-1-stimulated cells, including a reduction in the number and length of mitochondrial cristae. These cristae alterations were associated with reduced expression of CHCHD3 and CHCHD10, two proteins that form part of the mitochondrial contact site and cristae organizing system (MICOS). Although PD-1-stimulated cells showed severe cristae alterations, assembly of respiratory supercomplexes was unexpectedly greater in these cells than in activated T cells. CHCHD3 silencing in primary CD8+ T cells recapitulated some effects induced by PD-1 stimulation, including reduced mitochondrial polarization and interferon-γ production following T cell activation with anti-CD3 and -CD28 activating antibodies.
CONCLUSIONS: Our results suggest that mitochondria are the main targets of PD-1 inhibitory activity. PD-1 reprograms CD8+ T cell metabolism for efficient use of fatty acid oxidation; this mitochondrial phenotype might explain the long-lived phenotype of PD-1-engaged T cells.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.