Plant Cell Environ. 2024 Mar 18.
López B, Izquierdo Y, Cascón T, Zamarreño ÁM, García-Mina JM, Pulido P, Castresana C.
Abstract
Chloroplast function is essential for growth, development, and plant adaptation to stress. Organelle stress and plant defence responses were examined here using noxy8 (nonresponding to oxylipins 8) from a series of Arabidopsis mutants. The noxy8 mutation was located at the CLPC2 gene, encoding a chloroplast chaperone of the protease complex CLP. Although its CLPC1 paralogue is considered to generate redundancy, our data reveal significant differences distinguishing CLPC2 and CLPC1 functions. As such, clpc1 mutants displayed a major defect in housekeeping chloroplast proteostasis, leading to a pronounced reduction in growth and pigment levels, enhanced accumulation of chloroplast and cytosol chaperones, and resistance to fosmidomycin. Conversely, clpc2 mutants showed severe susceptibility to lincomycin inhibition of chloroplast translation and resistance to Antimycin A inhibition of mitochondrial respiration. In the response to Pseudomonas syringae pv. tomato, clpc2 but not clpc1 mutants were resistant to bacterial infection, showing higher salicylic acid levels, defence gene expression and 9-LOX pathway activation. Our findings suggest CLPC2 and CLPC1 functional specificity, with a preferential involvement of CLPC1 in housekeeping processes and of CLPC2 in stress responses.
DOI: 10.1111/pce.14882
Plant Cell Environ. 2022 Mar 15.
Noelia Arteaga, Belén Méndez-Vigo, Alberto Fuster-Pons, Marija Savic, Alba Murillo-Sánchez, F Xavier Picó, Carlos Alonso-Blanco
Abstract
Despite the adaptive and taxonomic relevance of the natural diversity for trichome patterning and morphology, the molecular and evolutionary mechanisms underlying these traits remain mostly unknown, particularly in organs other than leaves. In this study, we address the ecological, genetic and molecular bases of the natural variation for trichome patterning and branching in multiple organs of Arabidopsis (Arabidopsis thaliana). To this end, we characterized a collection of 191 accessions and carried out environmental and genome-wide association (GWA) analyses. Trichome amount in different organs correlated negatively with precipitation in distinct seasons, thus suggesting a precise fit between trichome patterning and climate throughout the Arabidopsis life cycle. In addition, GWA analyses showed small overlapping between the genes associated with different organs, indicating partly independent genetic bases for vegetative and reproductive phases. These analyses identified a complex locus on chromosome 2, where two adjacent MYB genes (ETC2 and TCL1) displayed differential effects on trichome patterning in several organs. Furthermore, analyses of transgenic lines carrying different natural alleles demonstrated that TCL1 accounts for the variation for trichome patterning in all organs, and for stem trichome branching. By contrast, two other MYB genes (TRY and GL1), mainly showed effects on trichome patterning or branching, respectively.
doi: 10.1111/pce.14308. Online ahead of print.
The Plant Cell, 4 Jan 2021, koaa041
Noelia Arteaga, Marija Savic, Belén Méndez-Vigo, Alberto Fuster-Pons, Rafael Torres-Pérez, Juan Carlos Oliveros, F Xavier Picó, Carlos Alonso-Blanco
Abstract
Both inter- and intra-specific diversity has been described for trichome patterning in fruits, which is presumably involved in plant adaptation. However, the mechanisms underlying this developmental trait have been hardly addressed. Here we examined natural populations of Arabidopsis (Arabidopsis thaliana) that develop trichomes in fruits and pedicels, phenotypes previously not reported in the Arabidopsis genus. Genetic analyses identified five loci, MALAMBRUNO 1–5 (MAU1–5), with MAU2, MAU3, and MAU5 showing strong epistatic interactions that are necessary and sufficient to display these traits. Functional characterization of these three loci revealed cis-regulatory mutations in TRICHOMELESS1 and TRIPTYCHON, as well as a structural mutation in GLABRA1. Therefore, the multiple mechanisms controlled by three MYB transcription factors of the core regulatory network for trichome patterning have jointly been modulated to trigger trichome development in fruits. Furthermore, analyses of worldwide accessions showed that these traits and mutations only occur in a highly differentiated relict lineage from the Iberian Peninsula. In addition, these traits and alleles were associated with low spring precipitation, which suggests that trichome development in fruits and pedicels might be involved in climatic adaptation. Thus, we show that the combination of synergistic mutations in a gene regulatory circuit has driven evolutionary innovations in fruit trichome patterning in Arabidopsis.
The EMBO Journal (2018) e99552
Cristina Martínez, Ana Espinosa‐Ruíz, Miguel de Lucas, Stella Bernardo‐García, José M Franco‐Zorrilla, Salomé Prat.
Abstract
The Arabidopsis PIF4 and BES1/BZR1 transcription factors antagonize light signaling by facilitating co‐activated expression of a large number of cell wall‐loosening and auxin‐related genes. While PIF4 directly activates expression of these targets, BES1 and BZR1 activity switch from a repressive to an activator function, depending on interaction with TOPLESS and other families of regulators including PIFs. However, the complexity of this regulation and its role in diurnal control of plant growth and brassinosteroid (BR) levels is little understood. We show by using a protein array that BES1, PIF4, and the BES1‐PIF4 complex recognize different DNA elements, thus revealing a distinctive cis‐regulatory code beneath BES1‐repressive and PIF4 co‐activation function. BES1 homodimers bind to conserved BRRE‐ and G‐box elements in the BR biosynthetic promoters and inhibit their expression during the day, while elevated PIF4 competes for BES1 homodimer formation, resulting in de‐repressed BR biosynthesis at dawn and in response to warmth. Our findings demonstrate a central role of PIF4 in BR synthesis activation, increased BR levels being essential to thermomorphogenic hypocotyl growth.
Nature Plants 2015; doi: 10.1038/nplants.2015.25.
Thieme CJ, Rojas-Triana M, Stecyk E, Schudoma C, Zhang W, Yang L, Miñambres M, Walther D, Schulze WX, Paz-Ares J, Scheible WR, Kragler F.
 The concept that proteins and small RNAs can move to and function in distant body parts is well established. However, non-cell-autonomy of small RNA molecules raises the question: To what extent are protein-coding messenger RNAs (mRNAs) exchanged between tissues in plants?
The concept that proteins and small RNAs can move to and function in distant body parts is well established. However, non-cell-autonomy of small RNA molecules raises the question: To what extent are protein-coding messenger RNAs (mRNAs) exchanged between tissues in plants?
Here we report the comprehensive identification of 2,006 genes producing mobile RNAs in Arabidopsis thaliana. The analysis of variant ecotype transcripts that were present in heterografted plants allowed the identification of mRNAs moving between various organs under normal or nutrient-limiting conditions. Most of these mobile transcripts seem to follow the phloem-dependent allocation pathway transporting sugars from photosynthetic tissues to roots via the vasculature. Notably, a high number of transcripts also move in the opposite, root-to-shoot direction and are transported to specific tissues including flowers. Proteomic data on grafted plants indicate the presence of proteins from mobile RNAs, allowing the possibility that they may be translated at their destination site. The mobility of a high number of mRNAs suggests that a postulated tissue-specific gene expression profile might not be predictive for the actual plant body part in which a transcript exerts its function.
Bio-protocol. 2014; 4 (20): e1261.
Chevalier F, Iglesias S M, Sánchez ÓJ, Montoliu L, Cubas P.
 The inflorescence stem of the flowering plant Arabidopsis thaliana (thale cress) is an excellent model system to investigate plant vascular tissue patterning and development. Plant vasculature is a complex conducting tissue arranged in strands called vascular bundles, formed by xylem (tissue that carries water) and phloem (tissue that carries photosynthates and signaling molecules). Xylem and phloem are originated from cell division of the meristematic cells of the vascular cambium. In Arabidopsis the flowering stem elongates about three weeks after germination. At this stage it is possible to visualize defects in its development and morphology.
The inflorescence stem of the flowering plant Arabidopsis thaliana (thale cress) is an excellent model system to investigate plant vascular tissue patterning and development. Plant vasculature is a complex conducting tissue arranged in strands called vascular bundles, formed by xylem (tissue that carries water) and phloem (tissue that carries photosynthates and signaling molecules). Xylem and phloem are originated from cell division of the meristematic cells of the vascular cambium. In Arabidopsis the flowering stem elongates about three weeks after germination. At this stage it is possible to visualize defects in its development and morphology.
Here we describe a protocol to embed in plastic (resin) stem segments either freshly dissected from living plants or previously assayed for β-glucuronidase. This protocol provides an excellent cellular morphology ideal to visualize stem cell types including those of vascular bundles using high-resolution light microscopy.
Plant Cell. 2014, pii: tpc.114.125047.
Chico JM, Fernández-Barbero G, Chini A, Fernández-Calvo P, Díez-Díaz M, Solano R.
 Reduction of the red/far-red (R/FR) light ratio that occurs in dense canopies promotes plant growth to outcompete neighbors but has a repressive effect on jasmonate (JA)–dependent defenses. The molecular mechanism underlying this trade-off is not well understood.
Reduction of the red/far-red (R/FR) light ratio that occurs in dense canopies promotes plant growth to outcompete neighbors but has a repressive effect on jasmonate (JA)–dependent defenses. The molecular mechanism underlying this trade-off is not well understood.
We found that the JA-related transcription factors MYC2, MYC3, and MYC4 are short-lived proteins degraded by the proteasome, and stabilized by JA and light, in Arabidopsis thaliana. Dark and CONSTITUTIVE PHOTOMORPHOGENIC1 destabilize MYC2, MYC3, and MYC4, whereas R and blue (B) lights stabilize them through the activation of the corresponding photoreceptors. Consistently, phytochrome B inactivation by monochromatic FR light or shade (FR-enriched light) destabilizes these three proteins and reduces their stabilization by JA. In contrast to MYCs, simulated shade conditions stabilize seven of their 10 JAZ repressors tested and reduce their degradation by JA.
MYC2, MYC3, and MYC4 are required for JA-mediated defenses against the necrotrophic pathogen Botrytis cinerea and for the shade-triggered increased susceptibility, indicating that this negative effect of shade on defense is likely mediated by shade-triggered inactivation of MYC2, MYC3, and MYC4. The opposite regulation of protein stability of MYCs and JAZs by FR-enriched light help explain (on the molecular level) the long-standing observation that canopy shade represses JA-mediated defenses, facilitating reallocation of resources from defense to growth.
Las bacterias que atacan a las plantas, además de secretar toxinas, inyectan en las células vegetales proteínas efectoras que favorecen la infección. Algunas cepas de Pseudomonas producen una toxina llamada coronatina que desorganiza la fisiología de la célula e impide el funcionamiento de sus defensas. La coronatina imita el funcionamiento de una de las hormonas encargadas de activar las defensas contra los hongos, el jasmonato. Pero, al mismo tiempo, promueve el crecimiento bacteriano al inhibir los mecanismos de defensa dependientes del ácido salicílico, los cuales son fundamentales para resitir las infecciones por Pseudomonas.
El grupo dirigido por Roberto Solano en el Centro Nacional de Biotecnología del CSIC intenta elucidar cómo las cepas de Pseudomonas que no producen coronatina, la mayoría de ellas, son capaces de alterar las hormonas de las plantas y qué mecanismos han evolucionado para que puedan infectarlas.
Aparte de las fitotoxinas, las cepas de Pseudomonas producen hasta 30 proteínas efectoras de tipo III distintas que son capaces de suprimir de forma activa las defensas de las plantas infectadas. Para el caso concreto de uno de estos efectores, HopX1, Solano acaba de encontar en su laboratorio del CNB que en la planta modelo Arabidopsis, tanto la expresión exógena de HopX1 como la infección natural por cepas de Pseudomonas que producen este efector, promueven el crecimeinto bacteriano de manera similar a la coronatina: activando la expresión de los genes dependientes del jasmonato. Sus resultados indican que HopX1 es una cisteína proteasa que imita los efectos de la coronatina al degradar a represores esenciales en el funcionamiento de la vía de señalización del jasmonato, las llamadas proteínas jasmonate-ZIM domain (JAZ).
Estos resultados, publicados en la revista PLoS Biology, muestran un mecanismo hasta ahora desconocido por el que los patógenos manipulan los puntos clave de la respuesta hormonal que usan las plantas para protegerse frente a las infecciones.
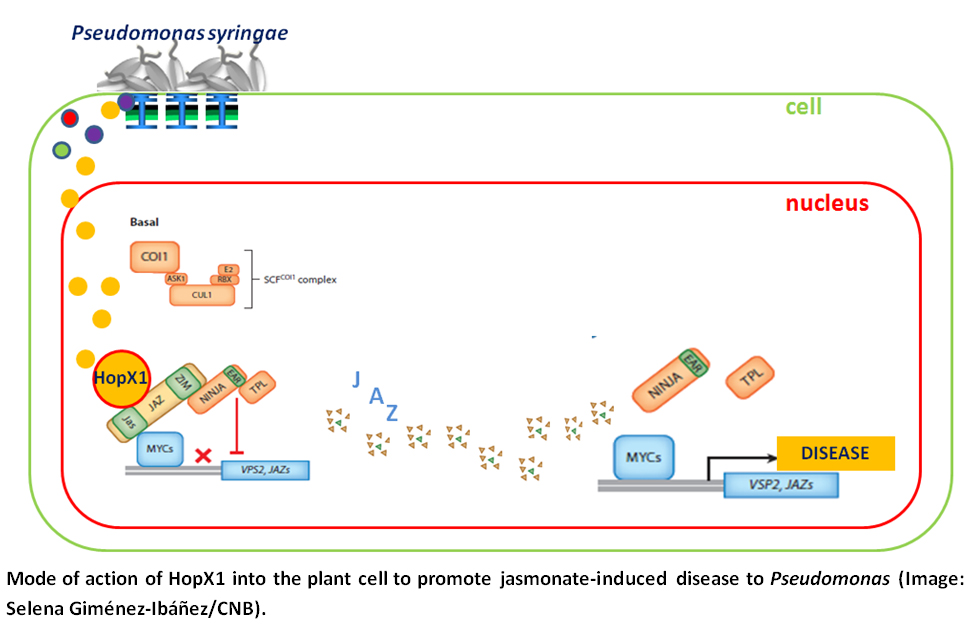
- Gimenez-Ibanez S, Boter M, Fernández-Barbero G, Chini A, Rathjen JP, Solano R. The bacterial effector HopX1 targets JAZ transcriptional repressors to activate jasmonate signaling and promote infection in Arabidopsis. PLoS Biol. 2014; 12(2): e1001792.
Plant Cell. 2014; 26 (2) :712-728.
Irigoyen ML1, Iniesto E, Rodriguez L, Puga MI, Yanagawa Y, Pick E, Strickland E, Paz-Ares J, Wei N, De Jaeger G, Rodriguez PL, Deng XW, Rubio V.
 CULLIN4-RING E3 ubiquitin ligases (CRL4s) regulate key developmental and stress responses in eukaryotes. Studies in both animals and plants have led to the identification of many CRL4 targets as well as specific regulatory mechanisms that modulate their function. The latter involve COP10-DET1-DDB1 (CDD)–related complexes, which have been proposed to facilitate target recognition by CRL4, although the molecular basis for this activity remains largely unknown.
CULLIN4-RING E3 ubiquitin ligases (CRL4s) regulate key developmental and stress responses in eukaryotes. Studies in both animals and plants have led to the identification of many CRL4 targets as well as specific regulatory mechanisms that modulate their function. The latter involve COP10-DET1-DDB1 (CDD)–related complexes, which have been proposed to facilitate target recognition by CRL4, although the molecular basis for this activity remains largely unknown.
Here, we provide evidence that Arabidopsis thaliana DET1-, DDB1-ASSOCIATED1 (DDA1), as part of the CDD complex, provides substrate specificity for CRL4 by interacting with ubiquitination targets. Thus, we show that DDA1 binds to the abscisic acid (ABA) receptor PYL8, as well as PYL4 and PYL9, in vivo and facilitates its proteasomal degradation. Accordingly, we found that DDA1 negatively regulates ABA-mediated developmental responses, including inhibition of seed germination, seedling establishment, and root growth. All other CDD components displayed a similar regulatory function, although they did not directly interact with PYL8. Interestingly, DDA1-mediated destabilization of PYL8 is counteracted by ABA, which protects PYL8 by limiting its polyubiquitination.
Altogether, our data establish a function for DDA1 as a substrate receptor for CRL4-CDD complexes and uncover a mechanism for the desensitization of ABA signaling based on the regulation of ABA receptor stability.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.