I. Fernández de Castro, R. Tenorio, P. Ortega-González, J.J. Knowlton, P.F. Zamora, C.H. Lee, J.J. Fernández, T.S. Dermody, C. Risco.
Abstract
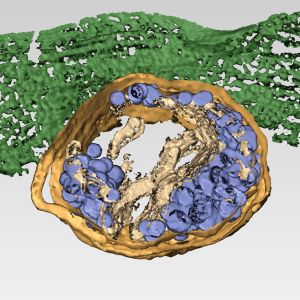
Mammalian orthoreoviruses (reoviruses) are nonenveloped viruses that replicate in cytoplasmic membranous organelles called viral inclusions (VIs) where progeny virions are assembled. To better understand cellular routes of nonlytic reovirus exit, we imaged sites of virus egress in infected, nonpolarized human brain microvascular endothelial cells (HBMECs) and observed one or two distinct egress zones per cell at the basal surface. Transmission electron microscopy and 3D electron tomography (ET) of the egress zones revealed clusters of virions within membrane-bound structures, which we term membranous carriers (MCs), approaching and fusing with the plasma membrane. These virion-containing MCs emerged from larger, LAMP-1-positive membranous organelles that are morphologically compatible with lysosomes. We call these structures sorting organelles (SOs). Reovirus infection induces an increase in the number and size of lysosomes and modifies the pH of these organelles from ∼4.5-5 to ∼6.1 after recruitment to VIs and before incorporation of virions. ET of VI-SO-MC interfaces demonstrated that these compartments are connected by membrane-fusion points, through which mature virions are transported. Collectively, our results show that reovirus uses a previously undescribed, membrane-engaged, nonlytic egress mechanism and highlights a potential new target for therapeutic intervention.
DOI: 10.1083/jcb.201910131.
- Es capaz de ‘apoderarse’ de los lisosomas celulares para dar salida a las partículas virales generadas en su interior
- Los factores de este mecanismo descrito por científicos del CSIC pueden llevar al diseño de nuevos fármacos antivirales
Un equipo liderado por investigadores del Consejo Superior de Investigaciones Científicas (CSIC) ha descrito el mecanismo utilizado por el reovirus humano, un patógeno causante de enfermedades respiratorias y digestivas en niños y jóvenes, para dar salida a las nuevas partículas virales fabricadas en el interior de las células infectadas. Este virus, que también se ha relacionado con el desarrollo de la celiaquía, es capaz de secuestrar los lisosomas celulares (unos orgánulos unidos a la membrana celular) para transportar las partículas virales infectivas desde el interior de la célula hasta la superficie. Los resultados, que podrían desembocar en el diseño de nuevos fármacos antivirales, aparecen publicados en la revista Journal of Cell Biology.
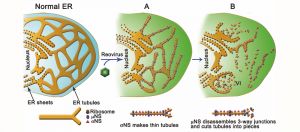
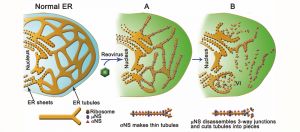
Los reovirus construyen orgánulos nuevos (factorías virales) en el citoplasma de las células infectadas para poder multiplicarse. En este trabajo se ha descubierto que el reovirus humano modifica y usa las membranas del retículo endoplásmico (RE) para formar sus orgánulos de replicación. Las proteínas no estructurales del reovirus, σNS y μNS, son las encargadas del remodelar el RE celular y de usarlo como andamiaje para ensamblar la factoría viral.
MBio. 2018 Aug 7;9(4). pii: e01253-18. doi: 10.1128/mBio.01253-18.
Tenorio R, Fernández de Castro I, Knowlton JJ, Zamora PF, Lee CH, Mainou BA, Dermody TS, Risco C.
Annu Rev Virol 2014; 1: 453-473.
Risco C, Fernández de Castro I, Sanz-Sánchez L, Narayan K, Grandinetti G, Subramaniam S.
 ReThree-dimensional (3D) imaging technologies are beginning to have significant impact in the field of virology, as they are helping us understand how viruses take control of cells.
ReThree-dimensional (3D) imaging technologies are beginning to have significant impact in the field of virology, as they are helping us understand how viruses take control of cells.
In this article we review several methodologies for 3D imaging of cells and show how these technologies are contributing to the study of viral infections and the characterization of specialized structures formed in virus-infected cells. We include 3D reconstruction by transmission electron microscopy (TEM) using serial sections, electron tomography, and focused ion beam scanning electron microscopy (FIB-SEM).
We summarize from these methods selected contributions to our understanding of viral entry, replication, morphogenesis, egress and propagation, and changes in the spatial architecture of virus-infected cells. In combination with live-cell imaging, correlative microscopy, and new techniques for molecular mapping in situ, the availability of these methods for 3D imaging is expected to provide deeper insights into understanding the structural and dynamic aspects of viral infection.
Los virus ARN, entre los que se encuentran muchos patógenos para humanos, animales y plantas, replican sus genomas en membranas del interior de las células. En estas membranas los virus reclutan factores celulares que participan en el ensamblaje y actividades de los complejos replicativos. Estos agregados macromoleculares, que suelen ensamblarse en vesículas con aperturas al citosol o “esférulas”, se encargan de fabricar múltiples copias del genoma viral que se incorporarán posteriormente en las nuevas partículas virales infectivas. Aunque se han identificado numerosos factores necesarios para el ensamblaje funcional de los orgánulos de replicación viral se desconoce la función exacta de la mayoría de ellos.
 En colaboración con investigadores de la Universidad de Kentucky en los Estados Unidos, el laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Cristina Risco han encontrado evidencias de una función inesperada y sorprendente que desempeña la proteína celular Vps4 en la replicación de un virus ARN perteneciente a la familia de los Tombusvirus, patógenos que infectan plantas y que causan importantes pérdidas en cosechas.
En colaboración con investigadores de la Universidad de Kentucky en los Estados Unidos, el laboratorio del Centro Nacional de Biotecnología del CSIC (CNB) dirigido por Cristina Risco han encontrado evidencias de una función inesperada y sorprendente que desempeña la proteína celular Vps4 en la replicación de un virus ARN perteneciente a la familia de los Tombusvirus, patógenos que infectan plantas y que causan importantes pérdidas en cosechas.
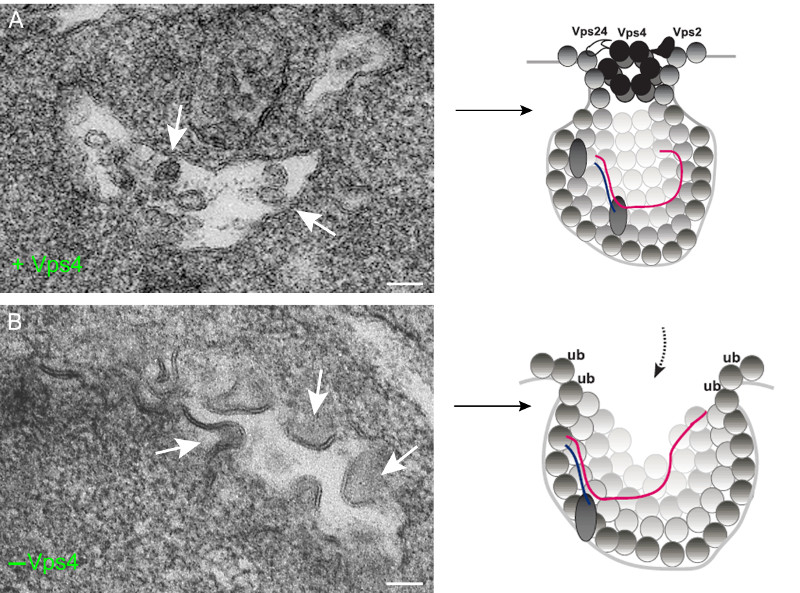
Vps4 es un componente de las proteínas celulares ESCRT y de la familia de las ATPasas AAA+ que usan ATP para remodelar estructuras macromoleculares en las células. Vps4 participa en una variedad de procesos biológicos como por ejemplo la fusión de membranas para la formación de vesículas. Utilizando nuevas técnicas de imagen en microscopía electrónica y según acaban de publicar en la revista PLoS Pathogens, los autores han demostrado que Vps4 participa en la estabilización del poro o cuello de las esférulas a través del cual se produce el intercambio de materiales con el citosol. Esta apertura controlada es necesaria para que el complejo replicativo desempeñe sus funciones y para que el ARN viral quede protegido de la degradación por nucleasas.
A diferencia de lo que ocurre en los procesos celulares habituales en los que participa, Vps4 se incorpora de manera estable en las vesículas virales o esférulas y pasa a formar parte del complejo replicativo. De hecho cuando Vps4 no está presente se forman esférulas totalmente abiertas, sin constricción o cuello y en las que el ARN viral de nueva síntesis está desprotegido. Los autores proponen que Vps4 y otras proteínas ESCRT son indispensables para la deformación de las membranas y el ensamblaje de los complejos replicativos de Tombusvirus. Es muy probable que otros virus de plantas y animales que transforman membranas celulares para construir esférulas también utilicen estas proteínas para ensamblar sus replicasas y proteger sus genomas recién sintetizados.
- Barajas D, Martín IFdC, Pogany J, Risco C, Nagy PD. Noncanonical role for the host Vps4 AAA+ ATPase ESCRT protein in the formation of Tomato bushy stunt virus replicase. PLoS Pathog. 2014; 10(4): e1004087.
PLoS Pathog 2014; 10(4): e1004087.
Barajas D, Martín IFdC, Pogany J, Risco C, Nagy PD.
Assembling of the membrane-bound viral replicase complexes (VRCs) consisting of viral- and host-encoded proteins is a key step during the replication of positive-stranded RNA viruses in the infected cells. Previous genome-wide screens with Tomato bushy stunt tombusvirus (TBSV) in a yeast model host have revealed the involvement of eleven cellular ESCRT (endosomal sorting complexes required for transport) proteins in viral replication. The ESCRT proteins are involved in endosomal sorting of cellular membrane proteins by forming multiprotein complexes, deforming membranes away from the cytosol and, ultimately, pinching off vesicles into the lumen of the endosomes.
In this paper, we show an unexpected key role for the conserved Vps4p AAA+ ATPase, whose canonical function is to disassemble the ESCRT complexes and recycle them from the membranes back to the cytosol. We find that the tombusvirus p33 replication protein interacts with Vps4p and three ESCRT-III proteins. Interestingly, Vps4p is recruited to become a permanent component of the VRCs as shown by co-purification assays and immuno-EM. Vps4p is co-localized with the viral dsRNA and contacts the viral (+)RNA in the intracellular membrane. Deletion of Vps4p in yeast leads to the formation of crescent-like membrane structures instead of the characteristic spherule and vesicle-like structures. The in vitro assembled tombusvirus replicase based on cell-free extracts (CFE) from vps4Δ yeast is highly nuclease sensitive, in contrast with the nuclease insensitive replicase in wt CFE.
These data suggest that the role of Vps4p and the ESCRT machinery is to aid building the membrane-bound VRCs, which become nuclease-insensitive to avoid the recognition by the host antiviral surveillance system and the destruction of the viral RNA. Other (+)RNA viruses of plants and animals might also subvert Vps4p and the ESCRT machinery for formation of VRCs, which require membrane deformation and spherule formation.
MBio. 2014; 5(1). pii: e00931-13.
Fernández de Castro I, Zamora PF, Ooms L, Fernández JJ, Lai CM, Mainou BA, Dermody TS, Risco C.
 Most viruses that replicate in the cytoplasm of host cells form neo-organelles that serve as sites of viral genome replication and particle assembly. These highly specialized structures concentrate viral replication proteins and nucleic acids, prevent the activation of cell-intrinsic defenses, and coordinate the release of progeny particles. Despite the importance of inclusion complexes in viral replication, there are key gaps in the knowledge of how these organelles form and mediate their functions.
Most viruses that replicate in the cytoplasm of host cells form neo-organelles that serve as sites of viral genome replication and particle assembly. These highly specialized structures concentrate viral replication proteins and nucleic acids, prevent the activation of cell-intrinsic defenses, and coordinate the release of progeny particles. Despite the importance of inclusion complexes in viral replication, there are key gaps in the knowledge of how these organelles form and mediate their functions.
Reoviruses are nonenveloped, double-stranded RNA (dsRNA) viruses that serve as tractable experimental models for studies of dsRNA virus replication and pathogenesis. Following reovirus entry into cells, replication occurs in large cytoplasmic structures termed inclusions that fill with progeny virions. Reovirus inclusions are nucleated by viral nonstructural proteins, which in turn recruit viral structural proteins for genome replication and particle assembly. Components of reovirus inclusions are poorly understood, but these structures are generally thought to be devoid of membranes. We used transmission electron microscopy and three-dimensional image reconstructions to visualize reovirus inclusions in infected cells.
These studies revealed that reovirus inclusions form within a membranous network. Viral inclusions contain filled and empty viral particles and microtubules and appose mitochondria and rough endoplasmic reticulum (RER). Immunofluorescence confocal microscopy analysis demonstrated that markers of the ER and ER-Golgi intermediate compartment (ERGIC) codistribute with inclusions during infection, as does dsRNA. dsRNA colocalizes with the viral protein σNS and an ERGIC marker inside inclusions. These findings suggest that cell membranes within reovirus inclusions form a scaffold to coordinate viral replication and assembly.
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.