Jaime Martín Benito
Jaime Martín Benito
ULTRAESTRUCTURA DE VIRUS Y AGREGADOS MOLECULARES
Nuestra principal línea de investigación es el estudio de la estructura de las ribonucleoproteínas (RNPs) del virus de la gripe A que son responsables de la transcripción y replicación viral .
Los plásmidos son moléculas de ADN circulares presentes en bacterias que se replican (duplican) de forma independiente del cromosoma bacteriano. La transmisión de plásmidos entre bacterias puede suponer la ganancia o la perdida de una función determinada en la célula receptora. Por ejemplo, algunos genes contenidos en plásmidos son capaces de conferir resistencia a antibióticos, y la transmisión de estos genes puede ser beneficiosa para la supervivencia bacteriana, ya que se convertirán en resistentes a esos antibióticos.
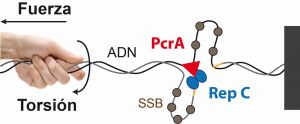
El sistema más habitual de duplicación de estos plásmidos se llama replicación por circulo rodante (RCR). Este proceso requiere la unión de la proteína Rep a una secuencia determinada del ADN, el corte de una de las cadenas del ADN y la separación de la doble cadena a medida que la helicasa PcrA se mueve por una de ellas. Sin embargo, el mecanismo por el cual se inicia la replicación y se desenrolla la doble hélice del ADN no se conoce con detalle.
Investigadores del Centro Nacional de Biotecnología, en colaboración con un grupo de científicos de la Universidad de Pittsburgh (Estados Unidos) han sido capaces de observar a nivel molecular la interacción de las proteínas Rep y PcrA en el proceso de replicación de plásmidos portadores de genes de resistencia a antibióticos.
Nucleic Acids Res. 2020 Jan 13. pii: gkz1200.
Carrasco C, Pastrana CL, Aicart-Ramos C, Leuba SH, Khan SA, Moreno-Herrero F.
Abstract
The rolling-circle replication is the most common mechanism for the replication of small plasmids carrying antibiotic resistance genes in Gram-positive bacteria. It is initiated by the binding and nicking of double-stranded origin of replication by a replication initiator protein (Rep). Duplex unwinding is then performed by the PcrA helicase, whose processivity is critically promoted by its interaction with Rep. How Rep and PcrA proteins interact to nick and unwind the duplex is not fully understood. Here, we have used magnetic tweezers to monitor PcrA helicase unwinding and its relationship with the nicking activity of Staphylococcus aureus plasmid pT181 initiator RepC. Our results indicate that PcrA is a highly processive helicase prone to stochastic pausing, resulting in average translocation rates of 30 bp s-1, while a typical velocity of 50 bp s-1 is found in the absence of pausing. Single-strand DNA binding protein did not affect PcrA translocation velocity but slightly increased its processivity. Analysis of the degree of DNA supercoiling required for RepC nicking, and the time between RepC nicking and DNA unwinding, suggests that RepC and PcrA form a protein complex on the DNA binding site before nicking. A comprehensive model that rationalizes these findings is presented.
doi: 10.1093/nar/gkz1200.
Nat Commun. 2020 Jan 2;11(1):55.
Vilas JL, Tagare HD, Vargas J, Carazo JM, Sorzano COS.
Abstract
The introduction of local resolution has enormously helped the understanding of cryo-EM maps. Still, for any given pixel it is a global, aggregated value, that makes impossible the individual analysis of the contribution of the different projection directions. We introduce MonoDir, a fully automatic, parameter-free method that, starting only from the final cryo-EM map, decomposes local resolution into the different projection directions, providing a detailed level of analysis of the final map. Many applications of directional local resolution are possible, and we concentrate here on map quality and validation.
doi: 10.1038/s41467-019-13742-w.
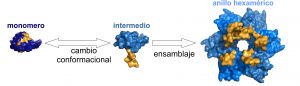
Muchas macromoléculas biológicas utilizan sistemas de control alostérico para ensamblarse, desensamblarse o cambiar de conformación. Así, cambios en una región de la molécula afectan al comportamiento y la función de la misma en otras regiones. Por ejemplo, la unión de un ligando produce un cambio conformacional que activa o inactiva una enzima.
El desarrollo de moléculas sintéticas que tengan este tipo de propiedades resulta muy complejo y hasta ahora los esfuerzos invertidos en este área solo han conseguido unos pocos resultados exitosos. De hecho, en el caso concreto de la creación de nuevos ensamblajes protéicos de diseño, los resultados hasta ahora se han basado en un sistema tipo puzzle, donde la superficie de cada una de las piezas rígidas que se van a ensamblar es modificada para producir una interacción estable. Ahora, investigadores del Centro Nacional de Biotecnología, en colaboración con el IMDEA Nanociencia, el Centro de Investigaciones Biológicas y las Universidades de Granada y California han desarrollado un modelo que permite controlar de forma alostérica la formación y ensamblaje de proteínas mediante la inclusión de interruptores moleculares formados gracias a cambios en la estructura tridimensional de la proteína.
Sachse M, Fernández de Castro I, Tenorio R, Risco C.
Abstract
Transmission electron microscopy (TEM) has been crucial to study viral infections. As a result of recent advances in light and electron microscopy, we are starting to be aware of the variety of structures that viruses assemble inside cells. Viruses often remodel cellular compartments to build their replication factories. Remarkably, viruses are also able to induce new membranes and new organelles. Here we revise the most relevant imaging technologies to study the biogenesis of viral replication organelles. Live cell microscopy, correlative light and electron microscopy, cryo-TEM, and three-dimensional imaging methods are unveiling how viruses manipulate cell organization. In particular, methods for molecular mapping in situ in two and three dimensions are revealing how macromolecular complexes build functional replication complexes inside infected cells. The combination of all these imaging approaches is uncovering the viral life cycle events with a detail never seen before.
doi: 10.1016/bs.aivir.2019.07.005.
Nucleic Acids Res. 2019 Aug 9.
Martín-González N, Hernando-Pérez M, Condezo GN, Pérez-Illana M, Šiber A, Reguera D, Ostapchuk P, Hearing P6, San Martín C, de Pablo PJ.
Abstract
Some viruses package dsDNA together with large amounts of positively charged proteins, thought to help condense the genome inside the capsid with no evidence. Further, this role is not clear because these viruses have typically lower packing fractions than viruses encapsidating naked dsDNA. In addition, it has recently been shown that the major adenovirus condensing protein (polypeptide VII) is dispensable for genome encapsidation. Here, we study the morphology and mechanics of adenovirus particles with (Ad5-wt) and without (Ad5-VII-) protein VII. Ad5-VII- particles are stiffer than Ad5-wt, but DNA-counterions revert this difference, indicating that VII screens repulsive DNA-DNA interactions. Consequently, its absence results in increased internal pressure. The core is slightly more ordered in the absence of VII and diffuses faster out of Ad5-VII- than Ad5-wt fractured particles. In Ad5-wt unpacked cores, dsDNA associates in bundles interspersed with VII-DNA clusters. These results indicate that protein VII condenses the adenovirus genome by combining direct clustering and promotion of bridging by other core proteins. This condensation modulates the virion internal pressure and DNA release from disrupted particles, which could be crucial to keep the genome protected inside the semi-disrupted capsid while traveling to the nuclear pore.
doi: 10.1093/nar/gkz687
POLÍTICA DE COOKIES
Una cookie es un archivo de texto que se almacena en el ordenador o dispositivo móvil mediante un servidor Web y tan solo ese servidor será capaz de recuperar o leer el contenido de la cookie y permiten al Sitio Web recordar preferencias de navegación y navegar de manera eficiente. Las cookies hacen la interacción entre el usuario y el sitio Web más rápida y fácil.
Información general
Está página Web utiliza cookies. Las cookies son pequeños archivos de texto generados por las páginas web que usted visita, las cuales contienen los datos de sesión que pueden ser de utilidad posteriormente en la página web. De esta forma esta Web recuerda información sobre su visita, lo que puede facilitar su próxima visita y hacer que el sitio Web le resulte más útil.
¿Cómo funcionan las cookies?
Las cookies sólo pueden almacenar texto, por lo general siempre es anónimo y cifrado. No se almacenarán información personal alguna en una cookie, ni pueden asociarse a persona identificada o identificable.
Los datos permiten que esta Web pueda mantener su información entre las páginas, y también para analizar la forma de interactuar con el sitio Web. Las cookies son seguras ya que sólo pueden almacenar la información que se puso en su lugar por el navegador, lo que es información que el usuario ha introducido en el navegador o la que se incluye en la solicitud de página. No puede ejecutar el código y no se puede utilizar para acceder a su ordenador. Si una página web cifra la información de la cookie, sólo la página web puede leer la información.
¿Qué tipos de cookies utilizamos?
Las cookies que utiliza esta página Web se pueden distinguir según los siguientes criterios:
1. Tipos de cookies según la entidad que las gestiona:
Según quien sea la entidad que gestione el equipo o dominio desde donde se envían las cookies y trate los datos que se obtengan, podemos distinguir:
- Cookies propias: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio gestionado por el propio editor y desde el que se presta el servicio solicitado por el usuario.
- Cookies de terceros: son aquellas que se envían al equipo terminal del usuario desde un equipo o dominio que no es gestionado por el editor, sino por otra entidad que trata los datos obtenidos través de las cookies.
En el caso de que las cookies sean instaladas desde un equipo o dominio gestionado por el propio editor pero la información que se recoja mediante éstas sea gestionada por un tercero, no pueden ser consideradas como cookies propias.
2. Tipos de cookies según el plazo de tiempo que permanecen activadas:
Según el plazo de tiempo que permanecen activadas en el equipo terminal podemos distinguir:
- Cookies de sesión: son un tipo de cookies diseñadas para recabar y almacenar datos mientras el usuario accede a una página web. Se suelen emplear para almacenar información que solo interesa conservar para la prestación del servicio solicitado por el usuario en una sola ocasión (p.e. una lista de productos adquiridos).
- Cookies persistentes: son un tipo de cookies en el que los datos siguen almacenados en el terminal y pueden ser accedidos y tratados durante un periodo definido por el responsable de la cookie, y que puede ir de unos minutos a varios años.
3. Tipos de cookies según su finalidad:
Según la finalidad para la que se traten los datos obtenidos a través de las cookies, podemos distinguir entre:
- Cookies técnicas: son aquellas que permiten al usuario la navegación a través de una página web, plataforma o aplicación y la utilización de las diferentes opciones o servicios que en ella existan como, por ejemplo, controlar el tráfico y la comunicación de datos, identificar la sesión, acceder a partes de acceso restringido, recordar los elementos que integran un pedido, realizar el proceso de compra de un pedido, realizar la solicitud de inscripción o participación en un evento, utilizar elementos de seguridad durante la navegación, almacenar contenidos para la difusión de vídeos o sonido o compartir contenidos a través de redes sociales.
- Cookies de personalización: son aquellas que permiten al usuario acceder al servicio con algunas características de carácter general predefinidas en función de una serie de criterios en el terminal del usuario como por ejemplo serian el idioma, el tipo de navegador a través del cual accede al servicio, la configuración regional desde donde accede al servicio, etc.
- Cookies de análisis: son aquellas que permiten al responsable de las mismas, el seguimiento y análisis del comportamiento de los usuarios de los sitios web a los que están vinculadas. La información recogida mediante este tipo de cookies se utiliza en la medición de la actividad de los sitios web, aplicación o plataforma y para la elaboración de perfiles de navegación de los usuarios de dichos sitios, aplicaciones y plataformas, con el fin de introducir mejoras en función del análisis de los datos de uso que hacen los usuarios del servicio.
Herramienta de gestión de las cookies
Está página Web utiliza Google Analytics.
Google Analytics es una herramienta gratuita de análisis web de Google que principalmente permite que los propietarios de sitios web conozcan cómo interactúan los usuarios con su sitio web. Asimismo, habilita cookies en el dominio del sitio en el que te encuentras y utiliza un conjunto de cookies denominadas "__utma" y "__utmz" para recopilar información de forma anónima y elaborar informes de tendencias de sitios web sin identificar a usuarios individuales.
Para realizar las estadísticas de uso de esta Web utilizamos las cookies con la finalidad de conocer el nivel de recurrencia de nuestros visitantes y los contenidos que resultan más interesantes. De esta manera podemos concentrar nuestros esfuerzos en mejorar las áreas más visitadas y hacer que el usuario encuentre más fácilmente lo que busca. En esta Web puede utilizarse la información de su visita para realizar evaluaciones y cálculos estadísticos sobre datos anónimos, así como para garantizar la continuidad del servicio o para realizar mejoras en sus sitios Web. Para más detalles, consulte en el siguiente enlace la política de privacidad [http://www.google.com/intl/es/policies/privacy/]
Cómo gestionar las cookies en su equipo: la desactivación y eliminación de las cookies
Todos los navegadores de Internet le permiten limitar el comportamiento de una cookie o desactivar las cookies dentro de la configuración o las opciones del navegador. Los pasos para hacerlo son diferentes para cada navegador, se pueden encontrar instrucciones en el menú de ayuda de su navegador.
Si no acepta el uso de las cookies, ya que es posible gracias a los menús de preferencias o ajustes de su navegador, rechazarlas, este sitio Web seguirá funcionando adecuadamente sin el uso de las mismas.
Puede usted permitir, bloquear o eliminar las cookies instaladas en su equipo mediante la configuración de las opciones del navegador instalado en su ordenador:
- Para más información sobre Internet Explorer pulse aquí.
- Para más información sobre Chrome pulse aquí.
- Para más información sobre Safari pulse aquí.
- Para más información sobre Firefox pulse aquí.
A través de su navegador, usted también puede ver las cookies que están en su ordenador, y borrarlas según crea conveniente. Las cookies son archivos de texto, los puede abrir y leer el contenido. Los datos dentro de ellos casi siempre están cifrados con una clave numérica que corresponde a una sesión en Internet por lo que muchas veces no tienen sentido más allá que la página web que los escribió.
Consentimiento informado
La utilización de la presente página Web por su parte, implica que Vd. presta su consentimiento expreso e inequívoco a la utilización de cookies, en los términos y condiciones previstos en esta Política de Cookies, sin perjuicio de las medidas de desactivación y eliminación de las cookies que Vd. pueda adoptar, y que se mencionan en el apartado anterior.